Перейти к:
Валидация и применение тест-системы на основе метода ПЦР в режиме реального времени для выявления ДНК Mycoplasma dispar
https://doi.org/10.29326/2304-196X-2025-14-3-274-282
Аннотация
Введение. В настоящее время наблюдается широкое распространение и активная циркуляция Mycoplasma dispar в животноводческих хозяйствах по всему миру, в том числе и в Российской Федерации. Актуальность внедрения в ветеринарную практику тест-системы на основе полимеразной цепной реакции в режиме реального времени для выявления ДНК Mycoplasma dispar обусловлена важной ролью данного возбудителя в развитии комплекса респираторных болезней крупного рогатого скота, приводящих к значительным экономическим потерям в животноводстве, в том числе являющихся причиной падежа телят.
Цель исследования. Определение основных валидационных характеристик и внедрение в ветеринарную практику разработанной тест-системы для выявления ДНК Mycoplasma dispar методом полимеразной цепной реакции в режиме реального времени.
Материалы и методы. Референтный штамм Mycoplasma dispar (ATCC № 27140) культивировали на питательной среде 1699 Revised Mycoplasma Medium, рекомендованной American Type Culture Collection. ДНК выделяли с использованием коммерческого набора, постановка ПЦР в режиме реального времени осуществлялась по заранее подобранным параметрам. Были определены основные валидационные характеристики тест-системы: аналитическая чувствительность, аналитическая специфичность, эффективность амплификации, повторяемость и воспроизводимость. Показана возможность практического применения тест-системы на основе ПЦР в режиме реального времени для выявления ДНК Mycoplasma dispar.
Результаты. Аналитическая чувствительность (предел обнаружения) тест-системы по выявлению ДНК Mycoplasma dispar составила 10 копий ДНК/мкл (или 100 копий ДНК/реакцию), специфичность – 100% (детектирует только ДНК Mycoplasma dispar), эффективность амплификации – 99,01%, среднее значение коэффициента вариации при оценке повторяемости – 0,91%, воспроизводимости – 0,66–1,26% в рамках 5 повторений и 0,91% в рамках 15 повторений. Апробацию тест-системы проводили на 228 пробах биоматериала, отобранных от крупного рогатого скота из 13 регионов Российской Федерации, при этом ДНК Mycoplasma dispar была обнаружена в 39,47% исследуемых проб.
Заключение. Разработанная тест-система для выявления ДНК Mycoplasma dispar продемонстрировала высокие валидационные показатели и может быть использована в диагностике микоплазмоза крупного рогатого скота.
Ключевые слова
Для цитирования:
Абед Алхуссен М., Федорова О.Е., Кротова А.О., Бьядовская О.П., Спрыгин А.В. Валидация и применение тест-системы на основе метода ПЦР в режиме реального времени для выявления ДНК Mycoplasma dispar. Ветеринария сегодня. 2025;14(3):274-282. https://doi.org/10.29326/2304-196X-2025-14-3-274-282
For citation:
Abed Alhussen M., Fedorova O.E., Krotova A.O., Byadovskaya O.P., Sprygin A.V. Validation and application of qPCR test kit for detection of Mycoplasma dispar DNA. Veterinary Science Today. 2025;14(3):274-282. https://doi.org/10.29326/2304-196X-2025-14-3-274-282
ВВЕДЕНИЕ
Mycoplasma dispar является одним из возбудителей респираторных заболеваний крупного рогатого скота (КРС), в том числе телят, и имеет повсеместное распространение. Болезнь, вызванная M. dispar, характеризуется воспалением слизистых оболочек верхних дыхательных путей и поражением легких, что приводит к значительным убыткам в животноводстве [1-3].
Данный вид микроорганизмов относится к роду Mycoplasma, классу Mollicutes, характеризуется отсутствием клеточной стенки, плеоморфизмом, небольшим для бактерий размером генома (803 т. п. н.) с низким соотношением G + C (28,5–29,3 моль%) [4][5].
Культивирование M. dispar требует специальной питательной среды для роста, микроорганизм не образует типичных для микоплазм колоний в виде «яичницы-глазуньи». Кроме того, M. dispar не всегда ингибируется гипериммунной сывороткой, что затрудняет традиционную идентификацию на ранних стадиях [5]. M. dispar ферментирует глюкозу и восстанавливает соли тетразолия в аэробных и анаэробных условиях, но не гидролизует аргинин, не катаболизирует сыворотку и не обладает фосфатазной активностью [6-8]. M. dispar имеет внешнюю капсулу, состоящую из полисахарида, идентифицированного как полимер галактуроновой кислоты [9]. Эта капсула образуется во время инфицирования клеток млекопитающих-хозяев и может оказывать ингибирующее действие на активность бычьих альвеолярных макрофагов, предотвращая их активацию [10].
Сообщалось, что для уничтожения микоплазмы альвеолярными макрофагами необходимо наличие антител против капсульного полисахарида [9].
Впервые М. dispar была выделена и идентифицирована в Англии в 1969 г. от телят с пневмонией. Затем данный вид микоплазм обнаружили в Дании, Бельгии, Голландии, Франции, Австралии, США, Канаде, Корее и Японии [8]. В Европе сообщения о выявлении возбудителя поступали из Великобритании, также инфекция, вызванная M. dispar, была зарегистрирована в Бразилии и Италии [11-15]. В Российской Федерации при исследовании 1186 проб биоматериала, полученных от КРС с клиническими признаками респираторной и/или репродуктивной патологии из 34 различных регионов в период с 2015 по 2018 г., методом полимеразной цепной реакции (ПЦР) с электрофоретической детекцией продуктов амплификации в агарозном геле ДНК М. dispar была обнаружена в 37,15% проб [16].
Передается M. dispar от инфицированных животных к здоровым в основном через дыхательные секреты, выявляется в дыхательных путях как здоровых, так и больных пневмонией телят [3]. При неблагоприятных условиях микоплазмы самостоятельно или в комбинации с другими инфекционными агентами могут вызывать серьезные респираторные заболевания, тем самым причиняя экономический ущерб крупным животноводческим хозяйствам с высокой концентрацией животных [17][18]. При обследовании поголовья КРС в Нидерландах M. dispar была выделена из 92% проб легких телят с пневмонией и из 40% проб легких здоровых телят [7][19]. В Дании при исследовании легких телят с признаками фибринозно-некротизирующей, гнойной бронхопневмонии и эмболической пневмонии M. dispar выявляли в 50% случаев [20]. Одновременно с микоплазмами обнаруживались и другие бактерии, такие как Histophilus somni, Pasteurella multocida, Arcanobacterium pyogenes и Mannheimia haemolytica. В Великобритании M. dispar часто выявляют у телят с пневмонией, считается, что она является причиной тяжелой плевропневмонии, сходной с контагиозной плевропневмонией КРС, включенной в список Всемирной организации здравоохранения животных (ВОЗЖ) и подлежащей обязательной нотификации [21].
Детальные исследования определили точную роль M. dispar в развитии комплекса респираторных заболеваний КРС, который является хронической болезнью, поражающей молочных телят и мясной скот на откормочных площадках. В результате было установлено, что в животноводческой отрасли США производственные потери, расходы на лечение и профилактику данного заболевания составляют более 4 млрд долларов [21].
Телятам с комплексом респираторных заболеваний обычно назначают антимикробные препараты, в то время как некоторые из них неэффективны против микоплазм [22]. Следовательно, существует значительная опасность возникновения резистентности к такого рода лекарственным средствам, что отмечается и при лечении респираторных инфекций, вызванных другими бактериальными патогенами [23].
Механизмы патогенности M. dispar достаточно изучены, они включают в себя способность вырабатывать перекись водорода и формировать биопленки – оба известных фактора вирулентности. Также было отмечено, что данный вид микоплазм колонизирует эпителий слизистой оболочки дыхательных путей, оказывая цитостатическое и даже цитопатическое действие на клетки дистальных бронхов и бронхиол и приводя к нарушению трахеобронхиального клиренса [9][21][24]. Кроме того, было показано, что M. dispar оказывает иммуносупрессивное действие на организм хозяина [25]. В связи с этим данный вид микоплазм был включен вместе с Mycoplasma bovis в число возбудителей, вызывающих или усугубляющих комплекс респираторных заболеваний [8][21]. M. dispar является причиной экссудативного бронхита и пневмонии у телят, передача инфекции происходит в основном воздушно-капельным путем и при тесном многократном контакте [26].
Mycoplasma dispar вызывает образование уплотнений пурпурно-красного цвета, в основном в краниовентральных областях легкого [27]. При экспериментальном заражении телят M. dispar развивались умеренный субклинический бронхиолит с лимфоидной инфильтрацией и легкая пневмония [10]; иногда отмечались случаи мастита [28]. Другие авторы сообщают, что M. dispar вызывает альвеолит, при котором нейтрофилы, макрофаги и экссудат накапливаются в альвеолярных стенках и пространствах [26][29]. При полевых случаях субклинической пневмонии, когда из проб биоматериала была выделена M. dispar, регистрировали сходные поражения [30].
Объем генетической информации по M. dispar в открытых базах данных ограничен. Полногеномная последовательность референтного штамма M. dispar ATCC № 27140 была внесена в GenBank в 2015 г. На основе филогенетического анализа последовательности гена 16S рРНК M. dispar сгруппирована вместе с M. ovipneumoniae, M. flocculare и M. hyopneumoniae, выделяемыми от овец и коз [31].
Борьба с микоплазмозом должна включать меры по снижению стресса, обусловленного условиями окружающей среды, и обеспечению надлежащего содержания животных в помещениях с хорошей циркуляцией воздуха. Необходимо проводить мероприятия, направленные на предотвращение заражения телят от взрослых животных [8].
Одним из ключевых компонентов борьбы с микоплазмозами КРС является своевременная диагностика. Лабораторное подтверждение заболевания, вызванного M. dispar, играет важную роль, в связи с глобальным распространением патогена, что оказывает значительное влияние на благополучие животноводческой отрасли [18][32][33][34].
Классическим способом идентификации микоплазм КРС является их выделение на питательных средах, что имеет приоритетное значение, так как в этом случае можно получить наиболее полную информацию о культурально-морфологических и биологических свойствах возбудителя. Кроме того, у данного метода имеется дополнительное преимущество, заключающееся в возможности создания банка клинических изолятов, которые в дальнейшем можно использовать при разработке и совершенствовании стратегий профилактики, контроля и искоренения микоплазмозов, а также для выявления резистентности микоплазм к антибактериальным препаратам [8][18][33][34]. Но следует также отметить, что данный способ трудоемок и требует 7–10 дней для постановки диагноза [3][18][34].
Серологические тесты на выявление антител к M. dispar широко не распространены, хотя в прошлом сообщалось о применении реакции радиального гемолиза, иммуноферментного анализа [35] и пассивной гемагглютинации [36, 37]. Авторами описано, что наличие антител к M. dispar у КРС слабо выражено, поскольку данный вид микоплазм обитает только на поверхности легких, однако это может быть связано с низкой чувствительностью используемого серологического анализа, а не с недостаточностью гуморального ответа организма животного [35].
Использование ПЦР значительно облегчило выявление микоплазм, хотя только в 2004 г. было сообщено о ПЦР, специфичной для выявления генома M. dispar [38]. В данной реакции применялись специфические и универсальные олигонуклеотиды для обнаружения единичных полиморфизмов в 16S рРНК последовательности. В то же время был описан метод ПЦР/ДГГЭ (денатурирующий градиентный гель-электрофорез), который позволяет выявлять и идентифицировать более 70 различных микоплазм, включая M. dispar [39][40].
В современных условиях оперативность диагностики может быть достигнута за счет применения ПЦР в режиме реального времени (ПЦР-РВ), которая обеспечивает быструю и точную идентификацию специфических локусов генома микоплазм в пробах биологического материала [18][32][34][41].
Использование флуоресцентных (TaqMan) зондов, которые распознают конкретную последовательность-мишень в межпраймерном сегменте, позволяет достичь высокой специфичности исследования. В отличие от традиционных методов ПЦР, метод в реальном времени не требует дополнительных операций с образцом после амплификации, что снижает риск контаминации и приводит к сокращению времени проведения анализа и увеличению пропускной способности.
Включение в состав тест-систем ПЦР-PB внутреннего контрольного образца (ВКО) позволяет с высокой степенью достоверности интерпретировать полученные данные и исключить ложноотрицательные результаты из-за наличия в пробе ингибиторов ПЦР [42]. Таким образом, актуальной задачей для производителей современных тест-систем на основе ПЦР-РВ является использование ВКО, который позволяет контролировать как этап выделения нуклеиновой кислоты, так и этап амплификации [43-46].
Также стоит отметить, что в соответствии с требованиями ВОЗЖ в каждый ПЦР-тест рекомендуется включать BKO для контроля качества [47].
Наличие инструментов молекулярной идентификации геномов микоплазм, в том числе M. dispar, позволяет не только отслеживать эпизоотическую ситуацию в отечественных хозяйствах, но и осуществлять контроль как при ввозе животных, так и при использовании их в производственных целях [34][48].
На сегодняшний день в Российской Федерации отсутствуют коммерчески доступные тест-системы ПЦР-РВ отечественного производства, выявляющие ДНК М. dispar, в связи с чем разработка и внедрение в ветеринарную практику специфичной диагностической тест-системы на основе ПЦР-РВ является актуальной задачей [34].
Целью данной работы являлось определение основных валидационных характеристик и внедрение в ветеринарную практику разработанной в ФГБУ «ВНИИЗЖ» тест-системы для выявления ДНК M. dispar методом ПЦР-РВ.
МАТЕРИАЛЫ И МЕТОДЫ
Бактерии и вирусы. В работе использовали референтный штамм M. dispar (ATCC № 27140), полученный из коллекции штаммов микроорганизмов ФГБУ «ВНИИЗЖ», а также пробы биологического материала, отобранные от КРС различных возрастных групп (стабилизированная кровь; назальные и трахеальные смывы; кусочки легких, трахеи и лимфатических узлов; плевральная жидкость).
Для оценки аналитической специфичности разработанной тест-системы были использованы штаммы бактерий: M. bovis ATCC № 25523, M. bovigenitalium ATCC № 19852, M. bovis «Калуга 2020», ДНК Mycoplasma mycoides subsp. mycoides SC (MmmSC) Madugri-8 (ФГБНУ «Федеральный исследовательский центр вирусологии и микробиологии», Россия), Mycoplasma mycoides subsp. mycoides SC (MmmSC) «T1/44/ВНИИЗЖ», а также другие бактериальные и вирусные агенты, способные вызвать аналогичную патологию у КРС: Escherichia coli «ЕС-21», Mannheimia haemolytica «№ 1412», Pasteurella multocida «№ 1414», изолят M. bovigenitalium, изолят M. dispar, изолят M. bovis, вирус парагриппа-3 КРС «ВГНКИ-4», респираторно-синцитиальный вирус КРС «Вологда/2020», вирус вирусной диареи КРС «NADL-ВНИИЗЖ».
Культивирование M. dispar. Референтный штамм M. dispar (ATCC № 27140) культивировали на рекомендованной American Type Culture Collection питательной среде 1699 Revised Mycoplasma Medium, состоящей из бульона с сердечно-мозговым экстрактом, – 7,5 г; 10× сбалансированного солевого раствора Хенкса – 40,0 мл; 0,25%-го раствора фенолового красного – 10,0 мл; термически инактивированной свиной сыворотки – 200,0 мл; 5%-го гидролизата лактальбумина в 1× фосфатно-буферном растворе – 100,0 мл; дрожжевого экстракта – 20,0 мл и дистиллированной воды – 660,0 мл. Культивирование проводили в инкубаторе при температуре (37 ± 0,5) °С и 5% CO2 в течение 5 сут. После завершения инкубации наблюдалось помутнение в пробирках и изменение цвета среды (пожелтение), на плотной питательной среде формировались колонии, характерные для M. dispar. Определение биологической активности М. dispar проводили путем подсчета колониеобразующих единиц (КОЕ/мл) [49][50].
Выделение ДНК осуществляли с использованием коммерческого набора «Ампли Прайм РИБО-сорб» (ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора, Россия) в соответствии с инструкцией производителя.
Постановка ПЦР-РВ. Реакционная смесь для амплификации на одну реакцию содержала следующие компоненты: ПЦР-буфер-Б для Taq ДНК-полимеразы 10× («Синтол», Россия); SynTaq ДНК-полимераза с ингибирующими активность фермента антителами, 5 ЕД/мкл («Синтол», Россия); 25 мМ водный раствор хлорида магния MgCl2 («Синтол», Россия); 100 мМ водных растворов четырех дезоксинуклеозидтрифосфатов (дНТФ): дАТФ, дГТФ, дТТФ, дЦТФ (готовится общая смесь дНТФ и разводится водой, свободной от нуклеаз, до концентрации 10 мМ каждого дНТФ; Fermentas, Литва); прямой праймер (100 пмоль/мкл), обратный праймер (100 пмоль/мкл) и TaqMan-зонд (100 пмоль/мкл) для выявления участка гена, кодирующего 16S рРНК M. dispar («Синтол», Россия); прямой праймер (100 пмоль/мкл), обратный праймер (100 пмоль/мкл) и TaqMan-зонд (100 пмоль/мкл) для выявления искусственно синтезированного ВКО («Синтол», Россия) [51]. Полученный объем реакционной смеси доводили до 15 мкл водой деионизированной, свободной от нуклеаз («Евроген», Россия). После добавляли в подготовленные пробирки по 15 мкл реакционной смеси и по 10 мкл ДНК-матрицы исследуемых образцов. Амплификацию проводили в программируемом амплификаторе Rotor-Gene Q (QIAGEN, Германия).
В качестве положительного контрольного образца (ПКО) использовали плазмидную конструкцию, содержащую олигонуклеотидную последовательность участка генома (с начальной концентрацией 2 × 107 копий/мкл) и специфичный участок генома M. dispar (целевой фрагмент).
В качестве отрицательного контрольного образца (ОКО) и отрицательного контроля ПЦР (ОК) использовали воду деионизированную, свободную от нуклеаз («Евроген», Россия).
Валидация. Валидационные параметры тест-системы определяли согласно рекомендованному S. A. Bustin et al. руководству по публикации результатов разработки количественных ПЦР-РВ-протоколов [52].
Для установления специфичности разработанной ПЦР-РВ были протестированы образцы биологического материала, содержащие ДНК представителей рода Mycoplasma и нуклеиновые кислоты вирусов и бактерий, способных вызывать аналогичную патологию у КРС. Чувствительность тест-системы определяли, используя заведомо положительные пробы биологического материала, содержащие ДНК M. dispar.
Предел обнаружения ДНК M. dispar (аналитическую чувствительность) разработанной тест-системы устанавливали, применяя ПКО, включающий ДНК M. dispar с начальной концентрацией 2 × 107 копий/мкл, каждое разведение исследовалось в 5 повторностях.
Эффективность амплификации оценивали с помощью серии последовательных 10-кратных разведений положительного образца биоматериала, содержащего ДНК M. dispar, в 3 повторностях и вычисляли согласно формуле:
Е = (101/slope − 1) × 100%,
где slope – это значение наклона линейной области зависимости Ct от логарифма концентрации кДНК-матрицы.
Статистическую обработку полученных результатов осуществляли с помощью программы Microsoft Excel: рассчитывали средние значения и их стандартные отклонения (± SD), проводили регрессионный анализ и вычисляли коэффициенты вариации. Коэффициент вариации (CV) при оценке воспроизводимости и сходимости (повторяемости) не должен превышать 10%.
Анализ промежуточной прецизионности в условиях повторяемости (воспроизводимость) проводили с одним образцом, который тестировали в 5 повторностях в течение трех постановок ПЦР-РВ (n = 15).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Впервые в России в ФГБУ «ВНИИЗЖ» была разработана и валидировна тест-система на основе ПЦР-РВ с применением ВКО для выявления ДНК M. dispar.
Использование ВКО значительно повышает достоверность и точность результатов ПЦР, в том числе решает проблемы ингибирования. Комитет Американской ассоциации ветеринарных лабораторных диагностов (AAVLD) рекомендовал, чтобы все внедряемые молекулярные диагностические тесты, которые проходят процедуру валидации, включали стратегию контроля ингибирования реакции, основанную на внутренней валидации для тестируемого образца, целевого вида-хозяина и комбинации матриц проб [53].
Начальным этапом валидации тест-системы был подбор температурного профиля, при котором достигается максимальная аналитическая чувствительность и эффективность амплификации.
Оптимизация температурно-временного режима ПЦР-РВ проводилась с ранее выверенными концентрациями компонентов реакции для получения средних значений порогового цикла Сt. Результаты представлены в таблице 1.
Был определен следующий оптимальный температурно-временной режим ПЦР-РВ для выявления ДНК M. dispar: 5 мин при 95 °С (прогрев реакционной смеси), далее 40 циклов ПЦР, состоящих из денатурации ДНК в течение 15 с при 95 °С, отжига праймеров и элонгации кДНК в течение 60 с при 60 °С.
В результате проведенных испытаний продемонстрировано, что разработанная тест-система позволяет избежать ложноположительных результатов при тестировании с генетическим материалом бактерий – возбудителей респираторных заболеваний КРС. Показано, что тест-система обладает 100%-й аналитической специфичностью в отношении M. dispar (табл. 2), что выше, чем в исследовании J. B. W. J. Cornelissen et al., где специфичность составила 98,2% [54]. Олигонуклеотиды также верифицировались по базе данных BLAST NCBI, при этом значимой гомологии ни с одним из видов микоплазм не установлено, то есть олигонуклеотиды уникальны для генома M. dispar.
Эффективность амплификации при постановке реакции оценивали с помощью серии последовательных 10-кратных разведений положительного образца, содержащего ДНК M. dispar. На основе полученных средних значений порогового цикла каждого разведения эффективность амплификации (Е) составила 99,01%, что оказалось более значительным по сравнению с 97,49%-й эффективностью, продемонстрированной J. B. W. J. Cornelissen et al. при разработке одноплексной ПЦР [54]. Коэффициент детерминации (R2) при этом был равен 0,9832 (рис. 1).
Внутрилабораторную прецизионность в условиях воспроизводимости оценивали с учетом таких показателей, как «персонал», «время» и «оборудование». Испытания осуществляли с использованием биологического материала, содержащего и не содержащего ДНК M. dispar. Проведенные исследования показали, что результаты выявления ДНК M. dispar с использованием разработанной тест-системы полностью совпадают с ожидаемым результатом и не зависят от показателей «время», «персонал» и «оборудование».
Оценку промежуточной прецизионности в условиях повторяемости (сходимости) проводили с одним образцом, который тестировали в 5 повторностях в течение трех постановок ПЦР (n = 15).
Среднее значение порогового цикла (Ct) на протяжении трех постановок ПЦР-РВ варьировало от 26,74 до 27,36 с разбросом стандартного отклонения (SD) от 0,18 до 0,34. Коэффициент вариации составил 0,91% при максимальном допустимом значении 10%. При суммировании результатов трех постановок ПЦР-РВ среднее значение порогового цикла (Ct) и стандартного отклонения было равно 27,03 и ± 0,25 соответственно (табл. 3).
На следующем этапе определяли предел обнаружения (аналитическую чувствительность) ДНК M. dispar, выделенной из ряда 10-кратных последовательных разведений искусственно синтезированной последовательности ДНК, соответствующей специфичному участку в геноме M. dispar, с исходной активностью 2 × 107 копий ДНК/мкл. Каждое разведение исследовалось в 5 повторностях (табл. 4).
Установлено, что для разработанной тест-системы на основе ПЦР-РВ предел обнаружения ДНК M. dispar составляет 10 копий ДНК/мкл (100 копий ДНК/реакцию).
В исследованиях, проведенных L. McAuliffe et al., для дифференциации 32 видов микоплазм был использован денатурирующий градиентный гель-электрофорез (DGGE) продукта ПЦР 16S рибосомальной ДНК. Данный метод позволяет быстро идентифицировать многие виды микоплазм, для которых не существует специфичной ПЦР и которые выявляются с помощью культуральных и серологических тестов [39][40]. Однако артефакты, вносимые в ходе ПЦР или последующего DGGE-анализа, приводят к искажению результатов при использовании этого метода для количественного анализа α-разнообразия или относительного обилия оперативных таксономических единиц [55].
Некоторые исследователи для выявления M. dispar в пробах биологического материала использовали классическую ПЦР [31][38][56], однако ПЦР-РВ позволяет в режиме реального времени без использования гелей быстрее получить более точные результаты [57].
Анализ и интерпретация результатов. Рекомендуемые параметры ПЦР-анализа для каналов Green/FAM и Red/Cy5 тождественны: установка динамического фона, корректировка уклона, устранение выбросов на 10%, линейная шкала и пороговое значение, которое составляет 0,05 (рис. 2). Результаты интерпретируются на основании наличия или отсутствия пересечения кривой флуоресценции с пороговой линией, что соответствует наличию или отсутствию на каналах Green/FAM и Red/Cy5 значений пороговых циклов амплификации (Ct) в соответствующих графах таблиц результатов, отображаемых амплификатором.
Результат ПЦР-РВ считают достоверным при условии получения корректных результатов для отрицательного контроля ПЦР, отрицательного контрольного образца «ОКО» (контроль экстракции) и положительного контроля «M. dispar». Требования к контролям описаны в таблице 5.
Интерпретацию результатов для исследуемых образцов необходимо проводить согласно требованиям, представленным в таблице 6.
С использованием разработанной тест-системы исследовали 228 проб биоматериала, отобранного от КРС с клиническими признаками респираторной патологии, поступивших в ФГБУ «ВНИИЗЖ» в 2024 г. из 13 регионов РФ. Результаты представлены в таблице 7.
Установлено, что частота обнаружения ДНК M. dispar в образцах биоматериала составила 39,47%. Полученные данные свидетельствуют о достаточно широком распространении M. dispar среди КРС в российских хозяйствах в 2024 г.
Таблица 1
Значения порогового цикла Сt при оптимизации температурно-временного режима ПЦР-РВ для выявления ДНК M. dispar (n = 3)
Table 1
Threshold cycle (Ct) values of optimized real-time polymerase chain reaction temperature-time profile for M. dispar DNA detection (n = 3)
|
Стадия реакции |
Температура |
Продолжительность |
Количество циклов |
Среднее значение Ct ± SD |
|
1-й режим |
||||
|
Прогрев реакционной смеси |
95 °С |
5 мин |
1 |
25,46 ± 0,37 |
|
Денатурация |
95 °С |
15 с |
40 |
|
|
Отжиг праймеров и элонгация |
60 °C |
60 с (измерение флуоресценции Green/FAM, Red/Cy5) |
||
|
2-й режим |
||||
|
Прогрев реакционной смеси |
95 °С |
5 мин |
1 |
26,30 ± 0,86 |
|
Денатурация |
95 °С |
10 с |
40 |
|
|
Отжиг праймеров |
60 °С |
20 с (измерение флуоресценции Green/FAM, Red/Cy5) |
||
|
Элонгация |
72 °С |
20 с |
||
|
3-й режим |
||||
|
Прогрев реакционной смеси |
95 °С |
5 мин |
1 |
28,87 ± 0,15 |
|
Денатурация |
95 °С |
10 с |
45 |
|
|
Отжиг праймеров |
58 °С |
20 с (измерение флуоресценции Green/FAM, Red/Cy5) |
||
|
Элонгация |
72 °С |
20 с |
||
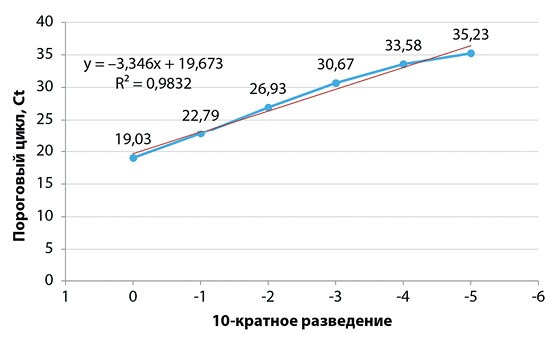
Рис. 1. Линейная зависимость результатов ПЦР-РВ при тестировании 10-кратных разведений ДНК генома M. dispar
Fig. 1. Linear correlation of real-time polymerase chain reaction results for 10-fold dilutions of the M. dispar genome DNA
Таблица 2
Определение специфичности разработанной тест-системы на основе ПЦР-РВ для выявления ДНК M. dispar (n = 3)
Table 2
Specificity assessment of the developed real-time polymerase chain reaction kit for M. dispar DNA detection (n = 3)
|
Генетический материал |
Штамм |
Результат ПЦР-РВ, Green/FAM (ДНК M. dispar) |
Результат ПЦР-РВ, Red/Cy5 (ДНК ВКО) |
|
Escherichia coli |
«ЕС-21» |
отр. |
+ |
|
Mannheimia haemolytica |
«№1412» |
отр. |
+ |
|
Pasteurella multocida |
«№1414» |
отр. |
+ |
|
Mycoplasma bovigenitalium |
ATCC № 19852 |
отр. |
+ |
|
Mycoplasma bovigenitalium |
изолят |
отр. |
+ |
|
Mycoplasma dispar |
ATCC № 27140 |
пол. |
+ |
|
Mycoplasma dispar |
изолят |
пол. |
+ |
|
Mycoplasma bovis |
ATCC № 25523 |
отр. |
+ |
|
Mycoplasma bovis |
«Калуга 2020» |
отр. |
+ |
|
Mycoplasma bovis |
изолят |
отр. |
+ |
|
Mycoplasma mycoides subsp. mycoides SC (MmmSC) |
«T1/44/ВНИИЗЖ» |
отр. |
+ |
|
Mycoplasma mycoides subsp. mycoides SC (MmmSC) |
Madugri-8 |
отр. |
+ |
|
Вирус парагриппа-3 КРС |
«ВГНКИ-4» |
отр. |
+ |
|
Респираторно-синцитиальный вирус КРС |
«Вологда/2020» |
отр. |
+ |
|
Вирус вирусной диареи КРС |
«NADL-ВНИИЗЖ» |
отр. |
+ |
|
Вода, свободная от нуклеаз |
– |
отр. |
+ |
|
отр. – ДНК M. dispar не выявлена (M. dispar DNA was not detected); пол. – ДНК M. dispar выявлена (M. dispar DNA was detected); «+» – ДНК ВКО выявлена (internal control sample DNA was detected). |
|||
Таблица 3
Вариабельность значений порогового цикла Ct в ПЦР-РВ для M. dispar (n = 15)
Table 3
Variability of real-time polymerase chain reaction Ct values for M. dispar (n = 15)
|
Номер постановки |
Номер повторности |
Полученное значение Ct |
Среднее значение Ct |
Стандартное отклонение (SD) |
Коэффициент вариации (Cv, %) |
|
I |
1 |
27,09 |
26,74 |
0,22 |
0,82 |
|
2 |
26,76 |
||||
|
3 |
26,51 |
||||
|
4 |
26,68 |
||||
|
5 |
26,64 |
||||
|
II |
1 |
27,41 |
27,36 |
0,18 |
0,66 |
|
2 |
27,50 |
||||
|
3 |
27,54 |
||||
|
4 |
27,13 |
||||
|
5 |
27,20 |
||||
|
III |
1 |
26,58 |
27,00 |
0,34 |
1,26 |
|
2 |
26,93 |
||||
|
3 |
27,32 |
||||
|
4 |
27,38 |
||||
|
5 |
26,77 |
||||
|
Итого |
27,03 |
0,25 |
0,91 |
||

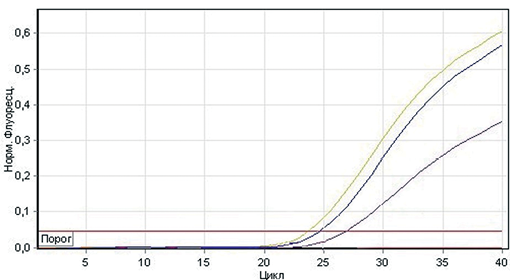
Рис. 2. Кривые флуоресценции: A – канал Green/FAM (ДНК M. dispar); B – канал Red/Cy5 (ДНК ВКО)
Fig. 2. Fluorescence curves: A – Green/FAM channel (M. dispar DNA); B – Red/Cy5 channel (internal control sample DNA)
Таблица 4
Аналитическая чувствительность (предел обнаружения) тест-системы ПЦР-РВ для выявления ДНК M. dispar
Table 4
Analytical sensitivity (detection limit) of the real-time polymerase chain reaction test kit for M. dispar DNA detection
|
Характеристика матрицы |
Результат выявления ДНК M. dispar, повторность |
Соответствие ожидаемому результату, % |
||||
|
1 |
2 |
3 |
4 |
5 |
||
|
2 × 107 копий ДНК/мкл |
пол. |
пол. |
пол. |
пол. |
пол. |
100 |
|
2 × 106 копий ДНК/мкл |
пол. |
пол. |
пол. |
пол. |
пол. |
100 |
|
2 × 105 копий ДНК/мкл |
пол. |
пол. |
пол. |
пол. |
пол. |
100 |
|
2 × 104 копий ДНК/мкл |
пол. |
пол. |
пол. |
пол. |
пол. |
100 |
|
2 × 103 копий ДНК/мкл |
пол. |
пол. |
пол. |
пол. |
пол. |
100 |
|
2 × 102 копий ДНК/мкл |
пол. |
пол. |
пол. |
пол. |
пол. |
100 |
|
20 копий ДНК/мкл |
пол. |
пол. |
пол. |
пол. |
пол. |
100 |
|
10 копий ДНК/мкл |
пол. |
пол. |
пол. |
пол. |
пол. |
100 |
|
5 копий ДНК/мкл |
отр. |
пол. |
отр. |
пол. |
отр. |
40 |
|
пол. – ДНК M. dispar выявлена (M. dispar DNA detected); |
||||||
Таблица 5
Требования к результатам контролей после амплификации
Table 5
Requirements for the control results following amplification
|
Контроль |
Значение порогового цикла (Ct) |
|
|
канал Green/FAM |
канал Red/Cy٥ |
|
|
Отрицательный контроль ПЦР |
отсутствует |
отсутствует |
|
Отрицательный контрольный образец «ОКО» |
отсутствует |
≤ 35 |
|
Положительный контроль «M. dispar» |
≤ 35 |
≤ 35 |
Таблица 6
Интерпретация результатов для исследуемых образцов
Table 6
Interpretation of the results for the tested samples
|
Значение порогового цикла (Ct) |
Результат |
|
|
канал Green/FAM (ДНК M. dispar) |
канал Red/Cy٥ (ВКО) |
|
|
отсутствует |
≤ 35 |
ДНК M. dispar не выявлена |
|
≤ 35 |
определено или отсутствует |
ДНК M. dispar выявлена |
|
> 35 |
≤ 35 |
сомнительный1 |
|
отсутствует или > 35 |
отсутствует или > 35 |
недостоверен2 |
|
1 провести повторное исследование для соответствующих образцов начиная с этапа экстракции ДНК, при повторении результата считать, что ДНК M. dispar в образце выявлена (re-test relevant samples, starting from the DNA extraction stage, if the test yields the same results, it can be assumed that M. dispar DNA has been detected in the original sample); |
||
Таблица 7
Идентификация и выявление генома M. dispar в пробах биоматериала с использованием разработанной тест-системы в 2024 г.
Table 7
Identification and detection of M. dispar genome in biological samples using the developed test kit in 2024
|
Регион |
Смывы (назальные, трахеальные) |
Патологический материал (стабилизированная кровь, кусочки легких, трахеи и лимфатических узлов, плевральная жидкость) |
||
|
Общее количество проб |
Количество положительных проб |
Общее количество проб |
Количество положительных проб |
|
|
Воронежская область |
– |
– |
52 |
0 |
|
Вологодская область |
– |
– |
2 |
0 |
|
Владимирская область |
12 |
10 |
24 |
4 |
|
Московская область |
22 |
8 |
2 |
2 |
|
Нижегородская область |
10 |
0 |
10 |
4 |
|
Чувашская Республика |
10 |
10 |
– |
– |
|
Краснодарский край |
– |
|
2 |
0 |
|
Республика Татарстан |
24 |
24 |
2 |
2 |
|
Рязанская область |
10 |
0 |
4 |
4 |
|
Ярославская область |
– |
– |
4 |
0 |
|
Республика Марий Эл |
– |
– |
2 |
2 |
|
Оренбургская область |
6 |
2 |
8 |
6 |
|
Самарская область |
16 |
6 |
6 |
6 |
|
Итого |
110 |
60 |
118 |
30 |
|
«–» – пробы для исследования из данного региона не поступали |
||||
ЗАКЛЮЧЕНИЕ
Таким образом, в результате проведенных исследований были установлены основные валидационные параметры и доказана возможность применения тест-системы «МИК-ДИСПАР ПЦР-РВ» (ФГБУ «ВНИИЗЖ») для выявления ДНК Mycoplasma dispar методом полимеразной цепной реакции в режиме реального времени, которая продемонстрировала высокую аналитическую специфичность и чувствительность, сходимость и воспроизводимость в различных условиях проведения теста (диапазон коэффициента вариации: 0,66–0,91%). Эффективность амплификации (E) составила 99,01%, а предел обнаружения – 100 копий ДНК/реакцию. Важно отметить, что данные показатели являются ключевыми в объективной оценке качества тест-систем ПЦР.
Использование экзогенного внутреннего контрольного образца позволяет исключить ложноотрицательные результаты. С применением разработанной тест-системы в 2024 г. ДНК M. dispar была выявлена в 39,47% исследованных проб, поступивших из разных регионов РФ в ФГБУ «ВНИИЗЖ», что свидетельствует об актуальности диагностических исследований на обнаружение ДНК M. dispar. При этом внутренний контрольный образец служит индикатором этапа выделения и присутствия возможных ингибиторов.
Следует отметить, что валидационная составляющая данного исследования может быть расширена за счет включения других родственных микоплазм, таких как M. bovirhinis, M. flocculare и М. ovipneumoniae, однако данные о выявлении указанных патогенов у крупного и мелкого рогатого скота на территории РФ отсутствуют. Более того, М. ovipneumoniae является возбудителем микоплазменной пневмонии овец и коз, и риски его циркуляции среди КРС ничтожно малы [58, 59, 60]. Учитывая высокую специфичность олигонуклеотидов при использовании ресурса BLAST, тест-система может применяться для диагностических исследований КРС на выявление ДНК M. dispar.
Таким образом, данная тест-система может служить эффективным инструментом в ветеринарной лабораторной практике для выявления ДНК M. dispar в пробах биологического материала от КРС.
Список литературы
1. Tortorelli G., Carrillo Gaeta N., Mendonça Ribeiro B. L., Miranda Marques L., Timenetsky J., Gregory L. Evaluation of Mollicutes microorganisms in respiratory disease of cattle and their relationship to clinical signs. Journal of Veterinary Internal Medicine. 2017; 31 (4): 1215–1220. https://doi.org/10.1111/jvim.14721
2. Chen S., Hao H., Yan X., LiuY., Chu Y. Genome-wide analysis of Mycoplasma dispar provides insights into putative virulence factors and phylogenetic relationships. G3: Genes, Genomes, Genetics. 2019; 9 (2): 317–325. https://doi.org/10.1534/g3.118.200941
3. Абед Алхуссен М., Кирпиченко В. В., Яцентюк С. П., Нестеров А. А., Бьядовская О. П., Жбанова Т. В., Спрыгин А. В. Патогенные микоплазмы крупного рогатого скота Mycoplasma bovis, M. bovigenitalium и M. dispar: краткая характеристика возбудителей (обзор). Сельскохозяйственная биология. 2021; 56 (2): 245–260. https://doi.org/10.15389/agrobiology.2021.2.245rus
4. Herrmann R. Genome structure and organization. In: Mycoplasmas: Molecular Biology and Pathogenesis. Ed. by J. Maniloff. Washington: American Society for Microbiology; 1992; 157–168.
5. Ter Laak E. A., Noordergraaf J. H. An improved method for the identification of Mycoplasma dispar. Veterinary Microbiology. 1987; 14 (1): 25–31. https://doi.org/10.1016/0378-1135(87)90049-6
6. Nicholas R. A. J., Khan L. A., Houshaymi B., Miles R. J., Ayling R. D., Hotzel H., Sachse K. Close genetic and phenotypic relatedness between Mycoplasma ovine/caprine serogroup 11 and Mycoplasma bovigenitalium. Systematic and Applied Microbiology. 2002; 25 (3): 396–402. https://doi.org/10.1078/0723-2020-00121
7. Ter Laak E. A., Noordergraaf J. H., Dieltjes R. P. J. W. Prevalence of mycoplasmas in the respiratory tracts of pneumonic calves. Journal of Veterinary Medicine, Series B. 1992; 39 (1–10): 553–562. https://doi.org/10.1111/j.1439-0450.1992.tb01205.x
8. Nicholas R., Ayling R., McAuliffe L. Mycoplasma diseases of ruminants. Wallingford: CABI; 2008. 239 p. https://doi.org/10.1079/9780851990125.0000
9. Almeida R. A., Rosenbusch R. F. Capsulelike surface material of Mycoplasma dispar induced by in vitro growth in culture with bovine cells is antigenically related to similar structures expressed in vivo. Infection and Immunity. 1991; 59 (9): 3119–3125. https://doi.org/10.1128/iai.59.9.3119-3125.1991
10. Howard C. J., Gourlay R. N., Taylor G. Immunity to Mycoplasma infections of the calf respiratory tract. Advances in Experimental Medicine and Biology. 1981; 137: 711–726. https://pubmed.ncbi.nlm.nih.gov/7331951
11. Ayling R. D., Bashiruddin S. E., Nicholas R. A. J. Mycoplasma species and related organisms isolated from ruminants in Britain between 1990 and 2000. Veterinary Record. 2004; 155 (14): 413–416. https://doi.org/10.1136/vr.155.14.413
12. França Dias de Oliveira B. A., Carrillo Gaeta N., Mendonça Ribeiro B. L., Reyes Alemán M. A., Miranda Marques L., Timenetsky J., et al. Determination of bacterial aetiologic factor on tracheobronchial lavage in relation to clinical signs of bovine respiratory disease. Journal of Medical Microbiology. 2016; 65 (10): 1137–1142. https://doi.org/10.1099/jmm.0.000345
13. Bottinelli M., Passamonti F., Rampacci E., Stefanetti V., Pochiero L., Coletti M., et al. DNA microarray assay and real-time PCR as useful tools for studying the respiratory tract Mycoplasma populations in young dairy calves. Journal of Medical Microbiology. 2017; 66 (9): 1342–1349. https://doi.org/10.1099/jmm.0.000571
14. Mosier D. Review of BRD pathogenesis: the old and the new. Animal Health Research Reviews. 2014; 15 (2): 166–168. https://doi.org/10.1017/S1466252314000176
15. Taylor G. Immunity to Mycoplasma infections of the respiratory tract: a review. Journal of the Royal Society of Medicine. 1979; 72 (7): 520–526. https://doi.org/10.1177/014107687907200711
16. Абед Алхуссен М., Нестеров А. А., Кирпиченко В. В., Яцентюк С. П., Спрыгин А. В., Бьядовская О. П., Кононов А. В. Распространение микоплазмозов крупного рогатого скота на животноводческих фермах в Российской Федерации в период с 2015 по 2018 год. Ветеринария сегодня. 2020; (2): 102–108. https://doi.org/10.29326/2304-196X-2020-2-33-102-108
17. Bernier Gosselin V., Francoz D., Babkine M., Desrochers A., Nichols S., Doré E, et al. A retrospective study of 29 cases of otitis media/interna in dairy calves. The Canadian Veterinary Journal. 2012; 53 (9): 957–962. https://pubmed.ncbi.nlm.nih.gov/23450859
18. Parker A. M., Sheehy P. A., Hazelton M. S., Bosward K. L., House J. K. A review of Mycoplasma diagnostics in cattle. Journal of Veterinary Internal Medicine. 2018; 32 (3): 1241–1252. https://doi.org/10.1111/jvim.15135
19. Ter Laak E. A., Noordergraaf J. H., Verschure M. H. Susceptibilities of Mycoplasma bovis, Mycoplasma dispar, and Ureaplasma diversum strains to antimicrobial agents in vitro. Antimicrobial Agents and Chemotherapy. 1993; 37 (2): 317–321. https://doi.org/10.1128/AAC.37.2.317
20. Tegtmeier C., Uttenthal A. A., Friis N. F., Jensen N. E., Jensen H. E. Pathological and microbiological studies on pneumonic lungs from Danish calves. Journal of Veterinary Medicine, Series B. 1999; 46 (10): 693–700. https://doi.org/10.1046/j.1439-0450.1999.00301.x
21. Bottinelli M., Merenda M., Gastaldelli M., Picchi M., Stefani E., Nicholas R. A. J., Catania S. The pathogen Mycoplasma dispar shows high minimum inhibitory concentrations for antimicrobials commonly used for bovine respiratory disease. Antibiotics. 2020; 9 (8):460. https://doi.org/10.3390/antibiotics9080460
22. DeDonder K. D., Apley M. D. A literature review of antimicrobial resistance in Pathogens associated with bovine respiratory disease. Animal Health Research Reviews. 2015; 16 (2): 125–134. https://doi.org/10.1017/S146625231500016X
23. Anholt R. M., Klima C., Allan N., Matheson-Bird H., Schatz C., Ajitkumar P., et al. Antimicrobial susceptibility of bacteria that cause bovine respiratory disease complex in Alberta, Canada. Frontiers in Veterinary Science. 2017; (4):207. https://doi.org/10.3389/FVETS.2017.00207
24. Dabo S. M., Taylor J. D., Confer A. W. Pasteurella multocida and bovine respiratory disease. Animal Health Research Reviews. 2007; 8 (2): 129–150. https://doi.org/10.1017/S1466252307001399
25. Almeida R. A., Wannemuehler M. J., Rosenbusch R. F. Interaction of Mycoplasma dispar with bovine alveolar macrophages. Infection and Immunity. 1992; 60 (7): 2914–2919. https://doi.org/10.1128/iai.60.7.2914-2919.1992
26. Friis N. F. Mycoplasma dispar as a causative agent in pneumonia of calves. Acta Veterinaria Scandinavica. 1980; 21: 34–42. https://doi.org/10.1186/bf03546898
27. Ross R. F. Mycoplasma – animal pathogens. In: Rapid Diagnosis of Mycoplasmas. Ed. by I. Kahane, A. Adoni. Boston: Springer; 1993; 69–109. https://doi.org/10.1007/978-1-4615-2478-6_7
28. Jasper D. E. Bovine mycoplasmal mastitis. Journal of the American Veterinary Medical Association. 1979; 175 (10): 1072–1074. https://doi.org/10.2460/javma.1979.175.10.1072
29. Gourlay R. N., Howard C. J., Thomas L. H., Wyld S. G. Pathogenicity of some Mycoplasma and Acholeplasma species in the lungs of gnotobiotic calves. Research in Veterinary Science. 1979; 27 (2): 233–237. https://doi.org/10.1016/S0034-5288(18)32836-4
30. George T. D., Horsfall N., Sullivan N. D. A subclinical pneumonia of calves associated with Mycoplasma dispar. Australian Veterinary Journal. 1973; 49 (12): 580–586. https://doi.org/10.1111/j.1751-0813.1973.tb06738.x
31. Shilan Faqe Muhammad Salih. Detection of Mycoplasma dispar in bovine respiratory disease by polymerase chain reaction assay in Sulaimaniyah city. Iraqi Journal of Agricultural Sciences. 2024; 55 (2): 703–710. https://doi.org/10.36103/p6042k98
32. Andersson A.-M., Aspán A., Wisselink H. J., Smid B., Ridley A., Pelkonen S., et al. A European inter-laboratory trial to evaluate the performance of three serological methods for diagnosis of Mycoplasma bovis infection in cattle using latent class analysis. BMC Veterinary Research. 2019; 15:369. https://doi.org/10.1186/s12917-019-2117-0
33. Dudek K., Nicholas R. A. J., Szacawa E., Bednarek D. Mycoplasma bovis infections – occurrence, diagnosis and control. Pathogens. 2020; 9 (8):640. https://doi.org/10.3390/pathogens9080640
34. Абед Алхуссен М. Молекулярно-биологические методы диагностики микоплазмозов крупного рогатого скота: дис. ... канд. вет. наук. Владимир: ФГБУ «ВНИИЗЖ»; 2023. 194 с.
35. Howard C. J. Mycoplasmas and bovine respiratory disease: studies related to pathogenicity and the immune response – a selective review. Yale Journal of Biology and Medicine. 1983; 56 (5–6): 789–797. https://pubmed.ncbi.nlm.nih.gov/6382831
36. Martin S. W., Bateman K. G., Shewen P. E., Rosendal S., Bohac J. E. The frequency, distribution and effects of antibodies, to seven putative respiratory pathogens, on respiratory disease and weight gain in feedlot calves in Ontario. Canadian Journal of Veterinary Research. 1989; 53 (3): 355–362. https://pubmed.ncbi.nlm.nih.gov/2766158
37. Martin S. W., Bateman K. G., Shewen P. E., Rosendal S., Bohac J. G., Thorburn M. A group level analysis of the associations between antibodies to seven putative pathogens and respiratory disease and weight gain in Ontario feedlot calves. Canadian Journal of Veterinary Research. 1990; 54 (3): 337–342. https://pubmed.ncbi.nlm.nih.gov/2165846
38. Miles K., McAuliffe L., Ayling R. D., Nicholas R. A. J. Rapid detection of Mycoplasma dispar and M. bovirhinis using allele specific polymerase chain reaction protocols. FEMS Microbiology Letters. 2004; 241 (1): 103–107. https://doi.org/10.1016/j.femsle.2004.10.010
39. McAuliffe L., Ellis R. J., Lawes J. R., Ayling R. D., Nicholas R. A. J. 16S rDNA PCR and denaturing gradient gel electrophoresis; a single generic test for detecting and differentiating Mycoplasma species. Journal of Medical Microbiology. 2005; 54 (8): 731–739. https://doi.org/10.1099/jmm.0.46058-0
40. McAuliffe L., Ellis R. J., Ayling R. D., Nicholas R. A. J. Differentiation of Mycoplasma species by 16S ribosomal DNA PCR and denaturing gradient gel electrophoresis fingerprinting. Journal of Clinical Microbiology. 2003; 41 (10): 4844–4847. https://doi.org/10.1128/JCM.41.10.4844-4847.2003
41. Baird S. C., Carman J., Dinsmore R. P., Walker R. L., Collins J. K. Detection and identification of Mycoplasma from bovine mastitis infections using a nested polymerase chain reaction. Journal of Veterinary Diagnostic Investigation. 1999; 11 (5): 432–435. https://doi.org/10.1177/104063879901100507
42. Moalic P.-Y., Gesbert F., Kempf I. Utility of an internal control for evaluation of a Mycoplasmameleagridis PCR test. Veterinary Microbiology. 1998; 61 (1–2): 41–49. https://doi.org/10.1016/S0378-1135(98)00173-4
43. Maaroufi Y., de Bruyne J.-M., Duchateau V., Scheen R., Crokaert F. Development of a multiple internal control for clinical diagnostic real-time amplification assays. FEMS Immunology and Medical Microbiology. 2006; 48 (2): 183–191. https://doi.org/10.1111/j.1574-695X.2006.00125.x
44. Hymas W., Stevenson J., Taggart E. W., Hillyard D. Use of lyophilized standards for the calibration of a newly developed real time PCR assay for human herpes type six (HHV6) variants A and B. Journal of Virological Methods. 2005; 128 (1–2): 143–150. https://doi.org/10.1016/j.jviromet.2005.05.003
45. Dingle K. E., Crook D., Jeffery K. Stable and noncompetitive RNA internal control for routine clinical diagnostic reverse transcription-PCR. Journal of Clinical Microbiology. 2004; 42 (3): 1003–1011. https://doi.org/10.1128/JCM.42.3.1003-1011.2004
46. Stöcher M., Leb V., Berg J. A convenient approach to the generation of multiple internal control DNA for a panel of real-time PCR assays. Journal of Virological Methods. 2003; 108 (1): 1–8. https://doi.org/10.1016/S0166-0934(02)00266-5
47. OIE Quality Standard and Guidelines for Veterinary Laboratories: Infectious Diseases. 2nd ed. 2008. 70 p.
48. Frey J., Nicolet J. Molecular identification and epidemiology of animal mycoplasmas. Wiener Klinische Wochenschrift. 1997; 109 (14–15): 600–603. https://pubmed.ncbi.nlm.nih.gov/9286067
49. Абед Алхуссен М., Нестеров А. А., Спрыгин А. В., Шумилова И. Н., Брянцева М. С., Бьядовская О. П. Оптимизация состава питательной среды и изучение стадий роста изолята «Калуга 2020» Mycoplasma bovis. Ветеринария сегодня. 2022; 11 (3): 262–267. https://doi.org/10.29326/2304-196X-2022-11-3-262-267
50. Hwang M.-H., Damte D., Cho M.-H., KimY.-H., Park S.-C. Optimization of culture media of pathogenic Mycoplasma hyopneumoniae by a response surface methodology. Journal of Veterinary Science. 2010; 11 (4): 327–332. https://doi.org/10.4142/jvs.2010.11.4.327
51. Морозова Е. О., Кротова А. О., Садчикова А. С., Тенитилов Н. А., Бьядовская О. П., Иголкин А. С. и др. Методические рекомендации по получению экзогенного внутреннего контрольного образца для ПЦР в режиме реального времени при выявлении ДНК микроорганизмов: утв. ФГБУ «ВНИИЗЖ» 13.10.2023 № 81-23. Владимир: ФГБУ «ВНИИЗЖ»; 2023. 32 с.
52. Bustin S. A., Benes V., Garson J. A., Hellemans J., Huggett J., Kubista M., et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clinical Chemistry. 2009; 55 (4): 611–622. https://doi.org/10.1373/clinchem.2008.112797
53. Yan L., Toohey-Kurth K. L., Crossley B. M., Bai J., Glaser A. L., Tallmadge R. L., Goodman L. B. Inhibition monitoring in veterinary molecular testing. Journal of Veterinary Diagnostic Investigation. 2020; 32 (6): 758–766. https://doi.org/10.1177/1040638719889315
54. Cornelissen J. B. W. J., de Bree F. M., van der Wal F. J., Kooi E. A., Koene M. G. J., Bossers A., et al. Mycoplasma detection by triplex real-time PCR in bronchoalveolar lavage fluid from bovine respiratory disease complex cases. BMC Veterinary Research. 2017; 13:97. https://doi.org/10.1186/s12917-017-1023-6
55. Neilson J. W., Jordan F. L., Maier R. M. Analysis of artifacts suggests DGGE should not be used for quantitative diversity analysis. Journal of Microbiological Methods. 2013; 92 (3): 256–263. https://doi.org/10.1016/j.mimet.2012.12.021
56. Marques L. M., Buzinhani M., Yamaguti M., Oliveira R. C., Ferreira J. B., Mettifogo E., Timenetsky J. Use of a polymerase chain reaction for detection of Mycoplasma dispar in the nasal mucus of calves. Journal of Veterinary Diagnostic Investigation. 2007; 19 (1): 103–106. https://doi.org/10.1177/104063870701900118
57. Zhang G., Brown E. W., González-Escalona N. Comparison of real-time PCR, reverse transcriptase real-time PCR, loop-mediated isothermal amplification, and the FDA conventional microbiological method for the detection of Salmonella spp. in produce. Applied and Environmental Microbiology. 2011; 77 (18): 6495–6501. https://doi.org/10.1128/AEM.00520-11
58. Deeney A. S., Collins R., Ridley A. M. Identification of Mycoplasma species and related organisms from ruminants in England and Wales during 2005–2019. BMC Veterinary Research. 2021; 17:325. https://doi.org/10.1186/s12917-021-03037-y
59. Chen J., Wang S., Dong D., Zhang Z., Huang Y., Zhang Y. Isolation and characterization of Mycoplasma ovipneumoniae infecting goats with pneumonia in Anhui Province, China. Life. 2024; 14 (2):218. https://doi.org/10.3390/life14020218
60. Wolfe L. L., Diamond B., Spraker T. R., Sirochman M. A., Walsh D. P., Machin C. M., et al. A bighorn sheep die-off in southern Colorado involving a Pasteurellaceae strain that may have originated from syntopic cattle. Journal of Wildlife Diseases. 2010; 46 (4): 1262–1268. https://doi.org/10.7589/0090-3558-46.4.1262
Об авторах
М. Абед АлхуссенРоссия
Абед Алхуссен Мохаммад - канд. вет. наук, младший научный сотрудник референтной лаборатории болезней крупного рогатого скота ФГБУ «ВНИИЗЖ».
ул. Гвардейская, 6, мкр. Юрьевец, Владимир, 600901
О. Е. Федорова
Россия
Федорова Ольга Евгеньевна - ведущий биолог референтной лаборатории болезней крупного рогатого скота ФГБУ «ВНИИЗЖ».
ул. Гвардейская, 6, мкр. Юрьевец, Владимир, 600901
А. О. Кротова
Россия
Кротова Алена Олеговна - ведущий биолог референтной лаборатории болезней крупного рогатого скота ФГБУ «ВНИИЗЖ».
ул. Гвардейская, 6, мкр. Юрьевец, Владимир, 600901
О. П. Бьядовская
Россия
Бьядовская Ольга Петровна - канд. биол. наук, заведующий референтной лаборатории болезней крупного рогатого скота ФГБУ «ВНИИЗЖ».
ул. Гвардейская, 6, мкр. Юрьевец, Владимир, 600901
А. В. Спрыгин
Россия
Спрыгин Александр Владимирович - д-р биол. наук, старший научный сотрудник референтной лаборатории болезней крупного рогатого скота ФГБУ «ВНИИЗЖ».
ул. Гвардейская, 6, мкр. Юрьевец, Владимир, 600901
Рецензия
Для цитирования:
Абед Алхуссен М., Федорова О.Е., Кротова А.О., Бьядовская О.П., Спрыгин А.В. Валидация и применение тест-системы на основе метода ПЦР в режиме реального времени для выявления ДНК Mycoplasma dispar. Ветеринария сегодня. 2025;14(3):274-282. https://doi.org/10.29326/2304-196X-2025-14-3-274-282
For citation:
Abed Alhussen M., Fedorova O.E., Krotova A.O., Byadovskaya O.P., Sprygin A.V. Validation and application of qPCR test kit for detection of Mycoplasma dispar DNA. Veterinary Science Today. 2025;14(3):274-282. https://doi.org/10.29326/2304-196X-2025-14-3-274-282
JATS XML



































