Перейти к:
Видовое разнообразие микроорганизмов и распространение антибиотикорезистентных энтеробактерий на молочных фермах
https://doi.org/10.29326/2304-196X-2025-14-3-294-301
Аннотация
Введение. Бактериальные сообщества существенно влияют на общую производительность сельскохозяйственных предприятий, от них зависит здоровье животных, производство молока, качество и безопасность пищевых продуктов. Зоонозные бактерии не только оказывают негативное воздействие на благополучие животных, но и представляют риск для общественного здравоохранения, поэтому мониторинг видового разнообразия микроорганизмов на молочных фермах для определения преобладающих видов возбудителей и профилей антибиотикорезистентности имеет важное значение.
Цель исследования. Изучение видового разнообразия бактериальных сообществ на молочной ферме и мониторинг распространения антибиотикорезистентности у изолятов Escherichia coli и Proteus mirabilis для своевременной разработки мер по сдерживанию распространения устойчивых к антибактериальным препаратам микроорганизмов.
Материалы и методы. Для достижения поставленной цели проводили идентификацию микроорганизмов методом MALDI-ToF масс-спектрометрии и определяли антибиотикочувствительность выделенных культур с помощью диско-диффузионного метода.
Результаты. Установлено видовое разнообразие микроорганизмов, выделенных из проб экссудата c поверхности ран конечностей крупного рогатого скота, фекалий и образцов корма. Преобладающими микроорганизмами оказались оппортунистические и патогенные Escherichia coli и Proteus mirabilis, для них определены профили антибиотикорезистентности. Один из изолятов Escherichia coli был мультирезистентным, только комбинация амоксициллина и клавулановой кислоты проявила эффективность в подавлении роста данной культуры. Большая доля изолятов Proteus mirabilis обладала устойчивостью к препаратам из группы фторхинолонов и чувствительностью ко всем остальным исследованным антибактериальным средствам.
Заключение. Отмечены факторы, влияющие на видовое разнообразие микроорганизмов в раневом экссудате, фекалиях и кормах. Определение профилей антибиотикорезистентности энтеробактерий позволит провести ротацию антибактериальных препаратов в исследованных животноводческих организациях.
Ключевые слова
Для цитирования:
Зубарева В.Д., Безбородова Н.А., Аминева П.Г., Кривоногова А.С., Соколова О.В., Шкуратова И.А., Исакова М.Н. Видовое разнообразие микроорганизмов и распространение антибиотикорезистентных энтеробактерий на молочных фермах. Ветеринария сегодня. 2025;14(3):294-301. https://doi.org/10.29326/2304-196X-2025-14-3-294-301
For citation:
Zubareva V.D., Bezborodova N.A., Amineva P.G., Krivonogova A.S., Sokolova O.V., Shkuratova I.A., Isakova M.N. Microbial species diversity and antibiotic-resistant Enterobacteriaceae spread on dairy farms. Veterinary Science Today. 2025;14(3):294-301. https://doi.org/10.29326/2304-196X-2025-14-3-294-301
ВВЕДЕНИЕ
Бактериальные сообщества, циркулирующие на молочных фермах, оказывают значительное влияние на безопасность пищевых продуктов, качество молочной продукции и здоровье животных [1]. Концепция «Единое здоровье» критически важна для понимания распространения резистентности к антибактериальным средствам, она подразумевает взаимодействие между людьми, животными и окружающей средой, что особенно актуально из-за общей природы устойчивых к противомикробным препаратам бактерий человека и животных [2]. В мире полмиллиарда человек заняты в сфере животноводства и напрямую подвергаются воздействию зоонозных микроорганизмов [3]. Остаточные количества противомикробных препаратов, антибиотикорезистентные патогены часто обнаруживаются в отходах животного происхождения и загрязняют почвенную среду и сточные воды [4]. Escherichia coli является резервуаром для многих генетических детерминант резистентности к антибиотикам, которые могут передаваться животным и людям через многочисленные пути: при контакте с животными или продуктами животного происхождения, а также с объектами окружающей среды [4]. Proteus mirabilis – условно-патогенный микроорганизм семейства Enterobacteriaceae, вызывающий воспалительные заболевания кожи, дыхательных путей, мочевыводящих путей и желудочно-кишечного тракта. После E. coli он является наиболее распространенной условно-патогенной и зоонозной бактерией, которую обнаруживают у различных животных, таких как куры, утки, черепахи, крупный рогатый скот и другие домашние животные [5]. P. mirabilis встречается в различных средах: сточных водах, почве и желудочно-кишечном тракте животных и человека [6]. Несоблюдение рекомендаций по назначению противомикробных препаратов в животноводстве способствует распространению антибиотикорезистентности.
Актуальность данной работы определяется тем, что изучение состава бактериальных сообществ, циркулирующих в животноводческих помещениях, позволит определить приоритетные микроорганизмы, влияющие на здоровье животных. Новизна исследований заключается в получении ранее неизвестных данных о составе микробиоты корма, раневого экссудата и фекалий крупного рогатого скота и выявлении степени распространения антибиотикорезистентности энтеробактерий в сельскохозяйственных организациях Свердловской области.
Цель исследования – изучение видового разнообразия бактериальных сообществ, а также проведение мониторинга распространения антибиотикорезистентных энтеробактерий (E. coli и P. mirabilis) на молочных фермах.
МАТЕРИАЛЫ И МЕТОДЫ
Исследования проводили в 2023–2024 гг. в отделе геномных исследований и селекции животных Уральского научно-исследовательского ветеринарного института – структурного подразделения ФГБНУ «Уральский федеральный аграрный научно-исследовательский центр Уральского отделения Российской академии наук» и лаборатории ООО «Кволити Мед» (г. Екатеринбург). Работа выполнена в 4 сельскохозяйственных организациях Свердловской области, занимающихся разведением крупного рогатого скота голштинской породы. Всего была отобрана 61 проба материала: экссудат с поверхностей ран конечностей (25), фекалии (22) и пробы корма (14).
Сбор проб фекалий от коров проводился зондом-тампоном в пробирки с модифицированной средой Кэри – Блэра, разработанной для транспортировки и сохранения жизнеспособности возбудителей кишечных инфекций (FecalSwabTM, Copan, Италия). Пробы остального биоматериала помещали в пробирки с транспортной средой Amies (eSwab®, Copan, Италия).
В лаборатории ООО «Кволити Мед» методом истощающего штриха 10 мкл суспензии биоматериала с помощью стерильной калиброванной петли высевали на питательные среды: колумбийский агар (Bio-Rad Laboratories, Inc., Франция) с 5% дефибринированной крови барана (E&O Laboratories Ltd., Шотландия); агар Плоскирева (ФБУН «Государственный научный центр прикладной микробиологии и биотехнологии», Россия); ГРМ-агар (ФБУН «Государственный научный центр прикладной микробиологии и биотехнологии», Россия); среда питательная хромогенная UriSelect 4 (Bio-Rad Laboratories, Inc., Франция); агар Сабуро с 2% глюкозы и хлорамфениколом (SIFIN diagnostics GmbH, Германия). Засеянные чашки Петри помещали в термостат при (37 ± 1) °С и инкубировали 24 ч.
Выросшие колонии идентифицировали методом MALDI-ToF масс-спектрометрии с помощью прибора Vitek® MS (bioMérieux, Франция). Для этого бактериальную массу наносили на спот слайда, покрывали 1 мкл матрицы (α-циано-3-гидроксикоричная кислота), высушивали при комнатной температуре и считывали прибором масс-спектры рибосомальных белков, сравнивая их с базой данных с помощью программного обеспечения MYLA® (bioMérieux, Франция). Определяли род и вид изолятов в биоматериале с полуколичественной и количественной характеристикой (КОЕ/грамм и КОЕ/образец).
Чувствительность к антибиотикам устанавливали по стандартной методике European Committee on Antimicrobial Susceptibility Testing (EUCAST) с использованием агара Мюллера – Хинтона (Bio-Rad Laboratories, Inc., Франция) и дисков (Bio-Rad Laboratories, Inc., Франция), пропитанных препаратами с определенной нагрузкой (табл.). Для считывания антибиотикограмм использовали автоматический анализатор ADAGIO (Bio-Rad Laboratories, Inc., Франция). При интерпретации категорий чувствительности применяли критерии EUCAST (E. A. Elshafiee, S. M. Nader, S. M. Dorgham, D. A. Hamza; версия 12.0, действующая с 01.01.2022). Несмотря на то что, согласно приказу Минсельхоза России от 18 ноября 2021 г. № 771, цефтазидим, цефепим, цефотаксим, цефтриаксон, цефоперазон запрещены к применению в ветеринарных целях, многие из данных антибактериальных веществ ранее входили в состав лекарственных средств, используемых при терапии воспалительных заболеваний крупного рогатого скота, в связи с чем указанные препараты также были включены при оценке профиля антибиотикорезистентности изолятов E. coli и P. mirabilis.
Таблица
Антибактериальные препараты для оценки антибиотикочувствительности микроорганизмов
Table
Antibacterial drugs used for assessing the antibiotic susceptibility of microorganisms
|
Наименование препарата |
Группа антибиотиков |
Концентрация активного вещества, мкг |
|
Цефиксим |
цефалоспорины III поколения |
5 |
|
Цефподоксим Цефтазидим |
цефалоспорины III поколения |
10 |
|
Цефепим Цефотаксим Цефуроксим Цефтриаксон |
цефалоспорины II, III, IV поколений |
30 |
|
Цефоперазон |
цефалоспорины III поколения |
75 |
|
Марбофлоксацин Энрофлоксацин Левофлоксацин Ципрофлоксацин Норфлоксацин |
фторхинолоны |
5 |
|
Гентамицин |
аминогликозиды |
10 |
|
Амоксициллин |
полусинтетические пенициллины |
30 |
|
Амоксициллин / клавулановая кислота |
полусинтетические пенициллины / ингибиторы бета-лактамаз |
20/10 |
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Всего методом MALDI-ToF идентифицировано 189 изолятов микроорганизмов.
В исследованных пробах раневого экссудата (n = 86) среди оппортунистических и патогенных микроорганизмов наибольшую долю занимали (рис. 1): P. mirabilis (9,30%), E. coli и Trueperella pyogenes (по 8,14%), Bacteroides pyogenes (6,98%), Aerococcus viridans и Prevotella melaninogenica (по 5,81%), Aeromonas hydrophila и Candida catenulata (по 4,65%), Bacillus altitudinis/pumilus, Mannheimia haemolytica, Streptococcus agalactiae (3,49%).
По данным S. C. Liegenfeld et al. [7], в составе микробиоты инфицированных ран наиболее часто обнаруживаются следующие микроорганизмы: грамотрицательные – Pseudomonas aeruginosa, Acinetobacter baumannii, Enterobacteriaceae, E. coli, Klebsiella pneumoniae, Serratia marcescens, Enterobacter spp., Proteus spp. и Bacteroides spp.; грамположительные – Staphylococcus aureus, Streptococcus spp., Enterococcus spp., Micrococcus spp., Corynebacterium spp., Streptococcus pyogenes, Corynebacterium diphtheriae и коагулазоотрицательные стафилококки. Таким образом, представленные авторами сведения о видовом разнообразии раневой микрофлоры отличаются от результатов нашего исследования.
Распространенность P. mirabilis в пищевых продуктах животного происхождения и побочных продуктах животноводства недостаточно изучена [8]. Вероятнее всего, обнаружение P. mirabilis, как и E. coli, в раневом экссудате связано с фекальным загрязнением поверхности ран. Бактерии вида T. pyogenes входят в состав микробиоты кожи и слизистых оболочек верхних дыхательных путей, желудочно-кишечного тракта, мочеполовых путей животных и являются условно-патогенными микроорганизмами. Они вызывают различные гнойные инфекции, такие как метрит, мастит, пневмония и абсцессы, которые наносят значительный экономический ущерб животноводству [9]. B. pyogenes – представитель микробиоты полости рта кошек и собак, укусы этих животных являются основными факторами риска заражения для человека. Данная бактерия может вызывать ряд воспалительных заболеваний, включая инфекции кожи и мягких тканей, остеомиелит, метрит и абсцессы печени [10][11]. Обнаруженная в ходе исследований анаэробная бактерия P. melaninogenica участвует в развитии и прогрессировании копытной гнили у крупного рогатого скота [12]. Представители филума Aeromonas вызывают заболевания, связанные с развитием диареи у поросят и свиней, жеребят и лошадей; аборты и репродуктивные заболевания кобыл, септические артриты жеребят, септицемию собак, маститы коров, полиартриты телят, при этом A. hydrophila был определен как единственный источник развития инфекции в ране у коровы [13]. S. agalactiae – микроорганизм, способный индуцировать хронический мастит у коров. Помимо этого, стрептококк данного вида может колонизировать желудочно-кишечный тракт молочных коров, а при контаминации фекалиями раневых поверхностей конечностей – быть причиной обнаружения данного микроорганизма в раневом экссудате [14].
Значительно меньшее видовое разнообразие оппортунистических и патогенных микроорганизмов идентифицировано в пробах фекалий крупного рогатого скота (n = 59): E. coli – 28,81%, Aspergillus niger complex – 15,25%, Bacillus licheniformis – 8,47%, P. mirabilis и Enterococcus hirae – по 6,78%, отмечено присутствие и других представителей грибковой микробиоты (рис. 2).
Сокращение разнообразия фекальной микробиоты может быть связано с увеличением концентрированных кормов в рационе [15]. Высококонцентрированная диета приводит к снижению соотношения ацетата к пропионату и pH в рубце, что негативно влияет на общее состояние здоровья и продуктивность крупного рогатого скота [16]. На этом фоне возникает дисбиоз, вследствие чего могут увеличиваться популяции определенных бактерий, таких как условно-патогенные E. coli [16], превалирование которых в фекальной микробиоте коров выявлено в наших исследованиях. Фекальное загрязнение E. coli окружающей среды увеличивает риск развития у коров колиформного мастита [17] и воспалительных заболеваний репродуктивного тракта [18].
В пробах корма: силоса, сенажа, комбикорма (n = 44) – преобладающими оппортунистическими и патогенными видами оказались: Bacillus altitudinis/pumilus – 9,09%, Aspergillus niger complex, Bacillus cereus, Candida krusei, Candida rugosa и P. mirabilis – по 6,82% (рис. 3).
Виды микроорганизмов, обычно обнаруживаемые в силосе, включают: Bacillus pumilus, B. licheniformis, B. coagulans, B. sphaericus и B. cereus. Появление спор B. cereus в силосе неизбежно при нарушении технологического процесса заготовки и хранения кормов. Споры B. cereus проходят через желудочно-кишечный тракт крупного рогатого скота без изменений и выделяются с фекалиями, во время доения из-за загрязнения поверхностей сосков могут переноситься в сырое молоко [19]. Наличие Candida spp., в частности C. krusei, в пробах корма – неблагоприятный показатель, поскольку данные микроскопические грибы могут вызывать микотический мастит у коров [20]. В нашем исследовании в пробах силоса обнаруживали A. niger, что может оказывать как положительное, так и отрицательное влияние на организм коров. Данный вид аспергилл продуцирует β-глюкозидазу, фермент, расщепляющий цианогенные гликозиды, токсичные для крупного рогатого скота, что снижает риск отравления цианидом и улучшает качество силоса [21]. В то же время аспергиллез крупного рогатого скота, особенно вызванный A. niger, может приводить к микотическим абортам и маститам [22]. Выявление в пробах корма патогенных микроорганизмов свидетельствует о необходимости разработки и реализации мер по улучшению технологических процессов заготовки, хранения и контроля качества кормов в исследованных животноводческих организациях.
Для определения антибиотикочувствительности были выбраны изоляты E. coli и P. mirabilis, так как эти микроорганизмы оказались преобладающими практически во всех видах исследованного биологического материала, кроме того, они имеют важное значение в индуцировании заболеваний как животных, так и человека.
У культур E. coli (n = 17) диско-диффузионным методом обнаружена резистентность к препаратам из групп фторхинолонов – 35,29% и цефалоспоринов – 11,76%. При этом к цефуроксиму 80,0% выделенных из фекалий изолятов эшерихий были чувствительны только при увеличении экспозиции. Наибольшая резистентность выявлена к амоксициллину – 41,18%, а к амоксициллину / клавулановой кислоте отмечается снижение устойчивости на 29,42%. Резистентность к гентамицину установлена у 17,65% изолятов (рис. 4).
Фенотип множественной лекарственной устойчивости – устойчивость хотя бы к одному агенту из трех или более химических классов антибиотиков – выявлена у одного изолята E. coli, эффективным в подавлении роста данного изолята оказалась комбинация амоксициллина и клавулановой кислоты. Среди патогенов со множественной лекарственной устойчивостью, широко распространенных на молочных фермах, особую обеспокоенность вызывает E. coli, поскольку некоторые штаммы могут вызывать пищевые инфекции у людей [4]. В процессе экспериментов R. Manishimwe et al. [23] у изолятов E. coli наблюдали наличие резистентности к тетрациклину (8,2%), цефтриаксону (56,8%), ципрофлоксацину (77,3%) и комбинации налидиксовой кислоты и ципрофлоксацина (54,5%), то есть частота встречаемости устойчивых изолятов E. coli значительно выше, чем в проведенном нами исследовании.
Все изоляты E. coli, выделенные из образцов раневого экссудата, за исключением одного, резистентного к амоксициллину, оказались чувствительными ко всем исследованным антибиотикам. Alharbi N. S. et al. выявлено, что более 50% изолятов E. coli, полученных из раневого отделяемого, были устойчивы к цефазолину, ампициллину, цефуроксиму, ципрофлоксацину, мезлоциллину, моксифлоксацину, пиперациллину и тетрациклину; 70% изолятов продуцировали бета-лактамазы расширенного спектра действия [24].
Резистентность к фторхинолонам наблюдали у 60,0% изолятов P. mirabilis (n = 10), выделенных из проб раневого экссудата (рис. 5). Все изоляты были восприимчивы к препаратам из группы цефалоспоринов, за исключением цефуроксима, к которому чувствительность проявили 80% изолятов только при увеличенной экспозиции. Устойчивость к амоксициллину и его комбинации с клавулановой кислотой наблюдали у 40% изолятов, полученных из фекалий и раневого экссудата; 40% изолятов, выделенных из раневого отделяемого, были резистентны к гентамицину. В целом стоит отметить, что препараты из группы цефалоспоринов эффективны в отношении P. mirabilis.
В проведенных исследованиях выявлено, что высокая распространенность у кур штаммов P. mirabilis, устойчивых к группе пенициллинов, цефалоспоринов и сульфаниламидов, является прямым следствием использования антимикробных препаратов в птицеводстве [25, 26]. Распространение антибиотикорезистентных P. mirabilis у продуктивных животных и в окружающей среде сельскохозяйственных предприятий является актуальной проблемой общественного здравоохранения. Ранее уже была описана передача P. mirabilis с фенотипом множественной лекарственной устойчивости от животных к человеку при употреблении зараженных пищевых продуктов или при тесном контакте с животными [8][25].
В ходе работы при помощи метода MALDI-ToF установлено видовое разнообразие бактерий, выделенных из проб экссудата с поверхности ран конечностей крупного рогатого скота, фекалий и проб корма. Полученные данные могут быть использованы на сельскохозяйственных предприятиях для назначения рациональной антибиотикотерапии как раневых инфекций, так и в случае возникновения других воспалительных заболеваний, связанных с данными возбудителями.
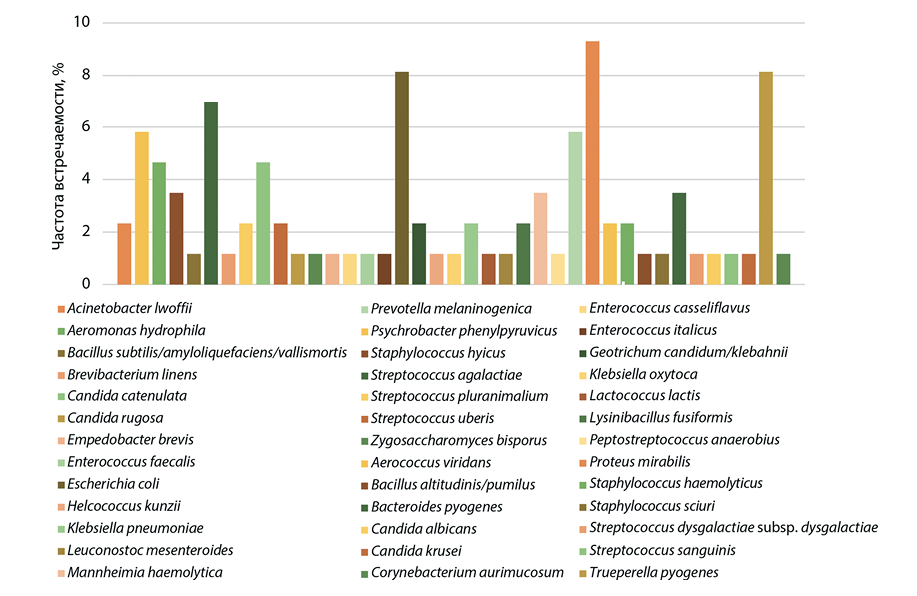
Рис. 1. Видовое разнообразие бактериальных сообществ, выделенных из раневого экссудата крупного рогатого скота (n = 86)
Fig. 1. Species diversity of bacterial communities isolated from wound exudates of cattle (n = 86)
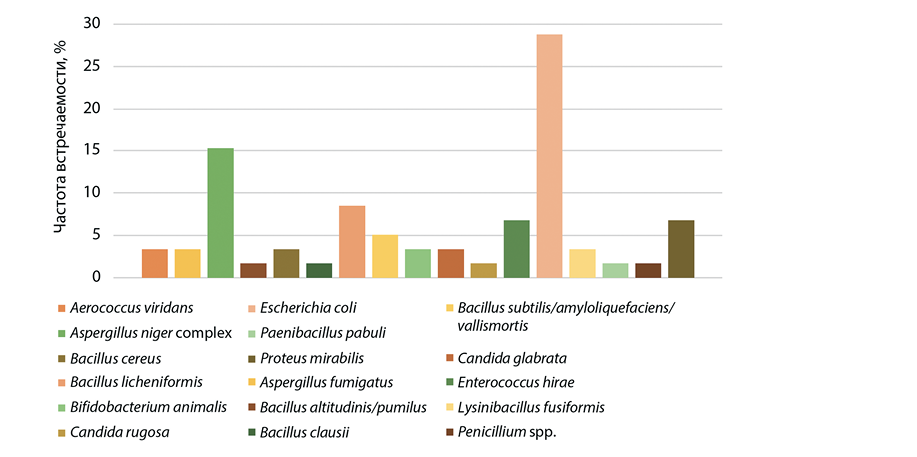
Рис. 2. Видовое разнообразие фекальной микробиоты крупного рогатого скота (n = 59)
Fig. 2. Fecal microbiota species diversity in cattle (n = 59)
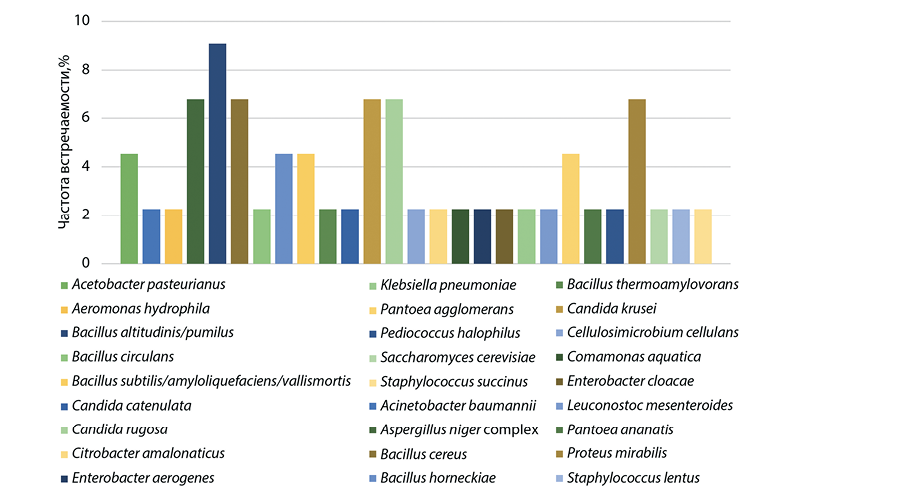
Рис. 3. Видовое разнообразие микроорганизмов, выделенных из проб корма (n = 44)
Fig. 3. Species diversity of microorganisms isolated from feed samples (n = 44)
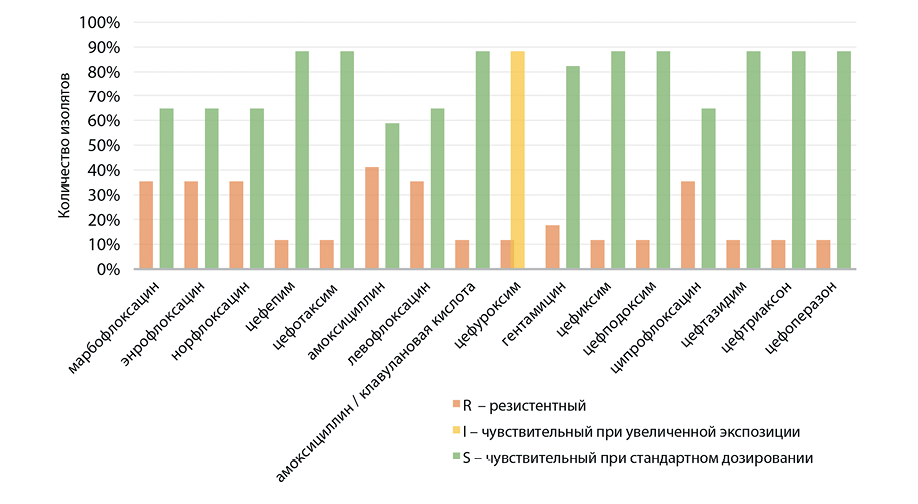
Рис. 4. Резистентность изолятов E. coli к антибактериальным препаратам (n = 17)
Fig. 4. Resistance of E. coli isolates to antibacterial drugs (n = 17)
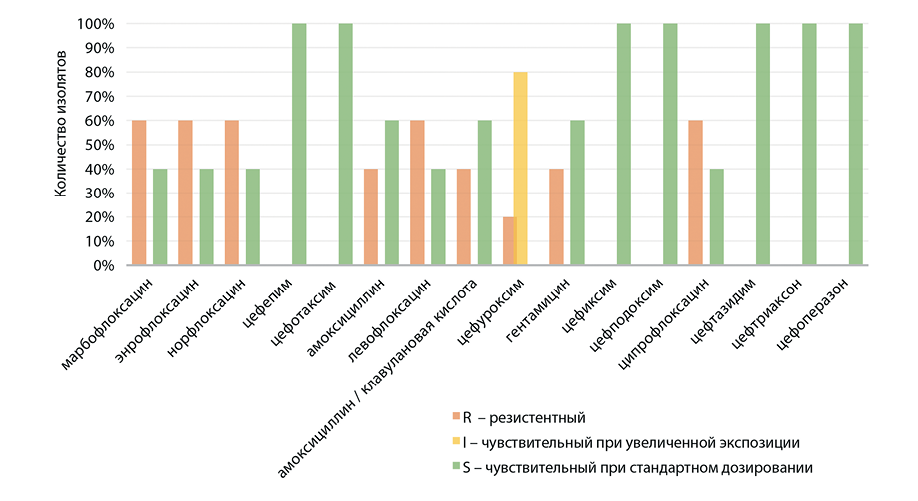
Рис. 5. Резистентность изолятов P. mirabilis к антибактериальным препаратам (n = 10)
Fig. 5. Resistance of P. mirabilis isolates to antibacterial drugs (n = 10)
ЗАКЛЮЧЕНИЕ
В результате проведенной работы в пробах раневого экссудата выявлено большое видовое разнообразие бактериальных сообществ, что в основном может быть связано с фекальной контаминацией ран конечностей. Фекальная же микробиота характеризовалась меньшим видовым составом, что может быть обусловлено возникновением дисбиоза вследствие увеличения доли концентрированных кормов в рационе коров, при этом в пробах фекалий установлено доминирование условно-патогенных E. coli (28,81%). Преобладание в пробах корма патогенных видов A. niger, B. cereus и C. krusei свидетельствует о необходимости изменения технологических процессов заготовки, хранения и осуществления контроля за качеством кормов в исследованных животноводческих организациях.
Установлены профили антибиотикорезистентности E. coli и P. mirabilis. У культур E. coli, изолированных из фекалий, устойчивость в основном обнаружена к препаратам из группы фторхинолонов (35,29%) и цефалоспоринов (11,76%). При этом практически все изоляты эшерихий, выделенные из раневого экссудата, оказались чувствительны ко всем исследованным антибактериальным препаратам. У одного изолята E. coli выявлен фенотип множественной лекарственной устойчивости, эффективным в подавлении его роста оказалась комбинация амоксициллина и клавулановой кислоты. Установлено, что практически все изоляты P. mirabilis были устойчивы к антибактериальным препаратам из группы фторхинолонов, эффективными в отношении P. mirabilis оказались препараты из группы цефалоспоринов. Для предотвращения дальнейшего роста антибиотикорезистентности необходимо проведение ротации антибактериальных препаратов, применяемых на молочных фермах, согласно выявленным профилям антибиотикорезистентности энтеробактерий.
Список литературы
1. Perdomo A., Calle A. Assessment of microbial communities in a dairy farm from a food safety perspective. International Journal of Food Microbiology. 2024; 423:110827. https://doi.org/10.1016/j.ijfoodmicro.2024.110827
2. Tyrrell C., Burgess C. M., Brennan F. P., Münzenmaier D., Drissner D., Leigh R. J., Walsh F. Genomic analysis of antimicrobial resistant Escherichiacoli isolated from manure and manured agricultural grasslands. NPJ Antimicrobials and Resistance. 2025; 3:8. https://doi.org/10.1038/s44259-025-00081-8
3. Mahmud B., Vargas R. C., Sukhum K. V., Patel S., Liao J., Hall L. R., et al. Longitudinal dynamics of farmer and livestock nasal and faecal microbiomes and resistomes. Nature Microbiology. 2024; 9: 1007–1020. https://doi.org/10.1038/s41564-024-01639-4
4. Veloo Y., Rajendiran S., Zakaria Z., Ismail R., Rahman S. A., Mansor R., Thahir S. S. A. Prevalence and antimicrobial resistance patterns of Escherichia coli in the environment, cow dung, and milk of Selangor dairy farms. Antibiotics. 2025; 14 (2):137. https://doi.org/10.3390/antibiotics14020137
5. Liu L., Dong Z., Ai S., Chen S., Dong M., Li Q., et al. Virulence-related factors and antimicrobial resistance in Proteus mirabilis isolated from domestic and stray dogs. Frontiers in Microbiology. 2023; 14:1141418. https://doi.org/10.3389/fmicb.2023.1141418
6. Al-Qurashi E., Elbnna K., Ahmad I., Abulreesh H. H. Antibiotic resistance in Proteus mirabilis: mechanism, status, and public health significance. Journal of Pure and Applied Microbiology. 2022; 16 (3): 1550–1561. https://doi.org/10.22207/JPAM.16.3.59
7. Liegenfeld S. C., Stenzel S., Rembe J.-D., Dittmer M., Ramos P., Stuermer E. K. Pathogenic and non-pathogenic microbes in the wound microbiome – how to flip the switch. Microbiology Research. 2025; 16 (2):39. https://doi.org/10.3390/microbiolres16020039
8. Chalmers G., Anderson R. E. V., Murray R., Topp E., Boerlin P. Characterization of Proteus mirabilis and associated plasmids isolated from anaerobic dairy cattle manure digesters. PloS ONE. 2023; 18 (8):e0289703. https://doi.org/10.1371/journal.pone.0289703
9. Rzewuska M., Kwiecień E., Chrobak-Chmiel D., Kizerwetter-Świda M., Stefańska I., Gieryńska M. Pathogenicity and virulence of Trueperella pyogenes: a review. International Journal of Molecular Sciences. 2019; 20 (11):2737. https://doi.org/10.3390/ijms20112737
10. Lee H. K., Walls G., Anderson G., Sullivan C., Wong C. A. Prolonged Bacteroides pyogenes infection in a patient with multiple lung abscesses. Respirology Case Reports. 2024; 12 (3):e01314. https://doi.org/10.1002/rcr2.1314
11. Cunha F., Jeon S. J., Jeong K. C., Galvão K. N. Draft genome sequences of Bacteroides pyogenes strains isolated from the uterus of Holstein dairy cows with metritis. Microbiology Resource Announcements. 2019; 8 (41):e01043-19. https://doi.org/10.1128/MRA.01043-19
12. Pyakurel S., Caddey B. J., Dias A. P., De Buck J., Morck D. W., Orsel K. Profiling bacterial communities in feedlot cattle affected with bovine foot rot and bovine digital dermatitis lesions using 16S rRNA gene sequencing and quantitative real-time PCR. BMC Microbiology. 2025; 25:158. https://doi.org/10.1186/s12866-025-03869-w
13. Awoyomi O. J., Oyewusi J. A., Talabi A. O., Oyewusi I. K., Biobaku K. T., Mustapha O. A., Agbaje M. Isolation of Aeromonas hydrophila in a case of wound infection in cattle in Nigeria. Nigerian Journal of Animal Production. 2014; 41 (1): 213–219. https://doi.org/10.51791/njap.v41i1.2726
14. Kabelitz T., Aubry E., van Vorst K., Amon T., Fulde M. The role of Streptococcus spp. in bovine mastitis. Microorganisms. 2021; 9 (7):1497. https://doi.org/10.3390/microorganisms9071497
15. Corrêa P. S., Jimenez C. R., Mendes L. W., Rymer C., Ray P., Gerdes L., et al. Taxonomy and functional diversity in the fecal microbiome of beef cattle reared in Brazilian traditional and semi-intensive production systems. Frontiers in Microbiology. 2021; 12:768480. https://doi.org/10.3389/fmicb.2021.768480
16. Auffret M. D., Dewhurst R. J., Duthie C. A., Rooke J. A., Wallace R. J., Freeman T. C., et al. The rumen microbiome as a reservoir of antimicrobial resistance and pathogenicity genes is directly affected by diet in beef cattle. Microbiome. 2017; 5:159. https://doi.org/10.1186/s40168-017-0378-z
17. Abegewi U. A., Esemu S. N., Ndip R. N., Ndip L. M. Prevalence and risk factors of coliform-associated mastitis and antibiotic resistance of coliforms from lactating dairy cows in North West Cameroon. PloS ОNE. 2022; 17 (7):e0268247. https://doi.org/10.1371/journal.pone.0268247
18. Yamamura F., Sugiura T., Munby M., Shiokura Y., Murata R., Nakamura T., et al. Relationship between Escherichia coli virulence factors, notably kpsMTII, and symptoms of clinical metritis and endometritis in dairy cows. Journal of Veterinary Medical Science. 2022; 84 (3): 420–428. https://doi.org/10.1292/jvms.21-0586
19. Driehuis F., Wilkinson J. M., Jiang Y., Ogunade I., Adesogan A. T. Silage review: Animal and human health risks from silage. Journal of Dairy Science. 2018; 101 (5): 4093–4110. https://doi.org/10.3168/jds.2017-13836
20. Elad D., Shpigel N. Y., Winkler M., Klinger I., Fuchs V., Saran A., Faingold D. Feed contamination with Candida krusei as a probable source of mycotic mastitis in dairy cows. Journal of the American Veterinary Medical Association. 1995; 207 (5): 620–622. https://pubmed.ncbi.nlm.nih.gov/7649779
21. Zhai J., Wang B., Sun Y., Yang J., Zhou J., Wang T., et al. Effects of Aspergillus niger on cyanogenic glycosides removal and fermentation qualities of ratooning sorghum. Frontiers in Microbiology. 2023; 14:1128057. https://doi.org/10.3389/fmicb.2023.1128057
22. Seyedmousavi S., Guillot J., Arné P., de Hoog G. S., Mouton J. W., Melchers W. J. G., Verweij P. E. Aspergillus and aspergilloses in wild and domestic animals: a global health concern with parallels to human disease. Medical Mycology. 2015; 53 (8): 765–797. https://doi.org/10.1093/mmy/myv067
23. Manishimwe R., Moncada P. M., Bugarel M., Scott H. M., Loneragan G. H. Antibiotic resistance among Escherichia coli and Salmonella isolated from dairy cattle feces in Texas. PloS ONE. 2021; 16 (5):e0242390. https://doi.org/10.1371/journal.pone.0242390
24. Alharbi N. S., Khaled J. M., Kadaikunnan S., Alobaidi A. S., Sharafaddin A. H., Alyahya S. A., et al. Prevalence of Escherichia coli strains resistance to antibiotics in wound infections and raw milk. Saudi Journal of Biological Sciences. 2019; 26 (7): 1557–1562. https://doi.org/10.1016/j.sjbs.2018.11.016
25. Sarwar A., Aslam B., Mahmood S., Muzammil S., Siddique A. B., Sarwar F., et al. Distribution of multidrug-resistant Proteusmirabilis in poultry, live-stock, fish, and the related environment: One Health heed. Veterinary World. 2025; 18 (2): 446–454. https://doi.org/10.14202/vetworld.2025.446-454
26. Кривоногова А. С., Донник И. М., Исаева А. Г., Логинов Е. А., Петропавловский М. В., Беспамятных Е. Н. Антибиотикорезистентность Enterobacteriaceae в микробиомах цыплят-бройлеров. Техника и технология пищевых производств. 2023; 53 (4): 710–717. https://doi.org/10.21603/2074-9414-2023-4-2472
Об авторах
В. Д. ЗубареваРоссия
Зубарева Владлена Дмитриевна - младший научный сотрудник отдела геномных исследований и селекции животных ФГБНУ УрФАНИЦ УрО РАН.
ул. Белинского, 112а, Екатеринбург, 620142
Н. А. Безбородова
Россия
Безбородова Наталья Александровна - канд. вет. наук, старший научный сотрудник, заведующий отделом геномных исследований и селекции животных ФГБНУ УрФАНИЦ УрО РАН.
ул. Белинского, 112а, Екатеринбург, 620142
П. Г. Аминева
Россия
Аминева Полина Геннадьевна - врач-микробиолог, заведующий лабораторией ООО «Кволити Мед».
ул. Машинная, 1, Екатеринбург, 620142
А. С. Кривоногова
Россия
Кривоногова Анна Сергеевна - д-р биол. наук, ведущий научный сотрудник отдела ветеринарно-лабораторной диагностики с испытательной лабораторией, ФГБНУ УрФАНИЦ УрО РАН.
ул. Белинского, 112а, Екатеринбург, 620142
О. В. Соколова
Россия
Соколова Ольга Васильевна - д-р вет. наук, ведущий научный сотрудник отдела геномных исследований и селекции животных, руководитель Уральского научно-исследовательского ветеринарного института – структурного подразделения ФГБНУ УрФАНИЦ УрО РАН.
ул. Белинского, 112а, Екатеринбург, 620142
И. А. Шкуратова
Россия
Шкуратова Ирина Алексеевна - д-р вет. наук, профессор, член-корреспондент РАН, главный научный сотрудник отдела экологии и незаразной патологии животных, ФГБНУ УрФАНИЦ УрО РАН.
ул. Белинского, 112а, Екатеринбург, 620142
М. Н. Исакова
Россия
Исакова Мария Николаевна - канд. вет. наук, старший научный сотрудник отдела репродуктивной биологии и неонатологии ФГБНУ УрФАНИЦ УрО РАН.
ул. Белинского, 112а, Екатеринбург, 620142
Рецензия
Для цитирования:
Зубарева В.Д., Безбородова Н.А., Аминева П.Г., Кривоногова А.С., Соколова О.В., Шкуратова И.А., Исакова М.Н. Видовое разнообразие микроорганизмов и распространение антибиотикорезистентных энтеробактерий на молочных фермах. Ветеринария сегодня. 2025;14(3):294-301. https://doi.org/10.29326/2304-196X-2025-14-3-294-301
For citation:
Zubareva V.D., Bezborodova N.A., Amineva P.G., Krivonogova A.S., Sokolova O.V., Shkuratova I.A., Isakova M.N. Microbial species diversity and antibiotic-resistant Enterobacteriaceae spread on dairy farms. Veterinary Science Today. 2025;14(3):294-301. https://doi.org/10.29326/2304-196X-2025-14-3-294-301
JATS XML



































