Перейти к:
Разработка и валидация высокочувствительного метода мультиплексной ОТ-ПЦР-РВ для обнаружения генома вируса классической чумы свиней
https://doi.org/10.29326/2304-196X-2024-13-3-223-233
Аннотация
Проблема классической чумы свиней в свиноводстве по-прежнему остается актуальной во всем в мире, несмотря на принимаемые меры. Последний случай данного заболевания в Российской Федерации регистрировали в 2020 г., однако сохраняется угроза эмерджентного возникновения болезни. Для предотвращения заноса вируса классической чумы свиней и быстрой ликвидации потенциально возможных вспышек необходимо проведение комплекса противоэпизоотических мероприятий, преимущественно включающих вакцинопрофилактику и ежегодный диагностический мониторинг на основе молекулярно-генетических и серологических исследований. В связи с этим разработан метод полимеразной цепной реакции с обратной транскрипцией в режиме реального времени с использованием внутреннего контрольного образца. Праймеры в модификации LockedNucleic Acid (конформационно блокированных нуклеозидов), обеспечивающие более высокий уровень аффинности к ДНК-матрице и физико-химической стабильности, и FAM-меченый TaqMan-зонд были подобраны к 5’-нетранслируемой области генома. Также определены валидационные показатели: правильность, сходимость, воспроизводимость, специфичность и чувствительность. С целью сравнительного анализа чувствительности параллельно тестировались зашифрованными тест-системами № х1, х2 образцы смывов, органов и тканей, полученных от свиней, экспериментально зараженных эпизоотическим штаммом вируса классической чумы свиней (селезенка, почка, печень, кровь, лимфатические узлы, ректальные и оральные мазки), корма, контаминированного животными, и вируссодержащего материала с известными титрами. Показаны 100%-я диагностическая чувствительность и предел детекции в 0,23 lg ККИД50/см3 разработанного метода. При этом показатели тест-систем № х1 и х2 были ниже, что может приводить к ложноотрицательным результатам полимеразной цепной реакции с обратной транскрипцией в режиме реального времени (ОТ-ПЦР-РВ) и влиять на недостоверную постановку диагноза. Таким образом, представленный метод может использоваться при проведении широкомасштабного мониторинга классической чумы свиней в Российской Федерации.
Ключевые слова
Для цитирования:
Садчикова А.С., Иголкин А.С., Чернышев Р.С., Козлов А.А., Колбин И.С., Спрыгин А.В., Бирюченков Д.А., Чвала И.А., Мазлум А. Разработка и валидация высокочувствительного метода мультиплексной ОТ-ПЦР-РВ для обнаружения генома вируса классической чумы свиней. Ветеринария сегодня. 2024;13(3):223-233. https://doi.org/10.29326/2304-196X-2024-13-3-223-233
For citation:
Sadchikova A.S., Igolkin A.S., Chernyshev R.S., Kozlov A.A., Kolbin I.S., Sprygin A.V., Biryuchenkov D.A., Chvala I.A., Mazloum A. Development and validation of highly sensitive multiplex real-time RT-PCR assay for detection of classical swine fever virus genome. Veterinary Science Today. 2024;13(3):223-233. https://doi.org/10.29326/2304-196X-2024-13-3-223-233
ВВЕДЕНИЕ
Классическая чума свиней (КЧС, Pestis suum) – одна из основных вирусных болезней, оказывающих значительное влияние на развитие свиноводства и охотничьего промысла дикого кабана [1].
Возбудителем КЧС является Pestivirus C рода Pestivirus семейства Flaviviridae, геном которого представлен одноцепочечной РНК положительной полярности длиной 12,3 т. н. [2]. Молекула РНК содержит две нетранслируемые области (5'-NTR и 3'-NTR), а также одну рамку считывания, кодирующую 13 белков (4 структурных и 9 неструктурных) [3].
Согласно рекомендациям Всемирной организации здравоохранения животных (ВОЗЖ), КЧС подлежит нотификации [4]. И несмотря на ликвидацию КЧС в европейских странах и России, где последняя вспышка регистрировалась в 2019 г. среди домашних свиней и в 2020 г. в популяции дикого кабана, угроза эмерджентного трансграничного заноса остается, что требует систематического надзора за заболеванием [5]. Ввиду отсутствия достоверных данных по количеству проведенного пассивного серологического мониторинга доказать циркуляцию вирулентного вируса КЧС в популяции дикого кабана на территории России затруднительно [6].
В настоящее время метод полимеразной цепной реакции (ПЦР) широко используется как один из самых быстрых, специфичных и чувствительных методов в молекулярной биологии, нацеленных на обнаружение генетического материала возбудителей болезней [7]. Однако метод классической ПЦР с электрофоретической детекцией в агарозном геле – долгий и трудоемкий, несущий высокий риск перекрестной контаминации [8]. Для диагностики КЧС, в том числе скринингового мониторинга, наиболее подходит мультиплексная полимеразная цепная реакция с обратной транскрипцией в режиме реального времени (ОТ-ПЦР-РВ) с внутренним контрольным образцом (ВКО), снижающим вероятность недостоверных результатов [9].
Зонды TaqMan, позволяющие проводить гибридизационно-флуоресцентную детекцию результатов ПЦР в режиме реального времени, как одни из самых практичных и надежных для диагностики пестивирусных инфекций, используются в ряде протоколов [10]. Большинство тест-систем на основе ОТ-ПЦР-РВ, описанных ранее для обнаружения генома вируса КЧС, амплифицируют фрагмент 5'-нетранслируемой области (5’-NTR) и демонстрируют приемлемую чувствительность и специфичность [11]. Более высокий уровень аффинности праймеров к ДНК-мишени и физико-химической стабильности достигается модификацией олигонуклеотидов в LNA (locked nucleic acid) [12].
Использование экзогенного ВКО дает возможность избегать ложноотрицательных результатов, возникающих вследствие ошибок как на стадии пробоподготовки (выделение нуклеиновой кислоты), так и амплификации целевого фрагмента [13].
Цель этого исследования заключалась в разработке и валидации высокочувствительного, специфичного и воспроизводимого протокола мультиплексной ОТ-ПЦР-РВ с ВКО, релевантного для диагностики всех субгенотипов вируса КЧС, циркулировавших на территории Российской Федерации (1.1, 1.2, 2.1, 2.3) в период с 1982 по 2020 г. [5][14]. Такой метод должен обладать характеристиками, удовлетворяющими всем требованиям для тест-системы на основе ПЦР-РВ, и возможностью широкого применения в диагностическом мониторинге инфекции [15].
МАТЕРИАЛЫ И МЕТОДЫ
Вирусы и бактерии. Для разработки методики использовали разные штаммы вируса КЧС: референтный «Ши-мынь», «684», «719» (субгенотип 1.1), вакцинный «СК» (субгенотип 1.2), «CSF Amur 19-10/WB-12555» и «CSF Tigrovoe 16/WB-634» (субгенотип 2.1), «275» (субгенотип 2.2), «368», «870», «843» (субгенотип 2.3) и штаммы с неопределенным генотипом «589», «924», «925», «929», «917», «918», «920», «926», «927», «930» вируса КЧС, выделенные в России в период с 1982 по 2020 г.
Для определения аналитической специфичности метода использовали гетерологичные возбудители болезней свиней: вакцинный штамм «ВК»-ДЕП вируса болезни Ауески; штамм «Иркутский 2007» американского вируса репродуктивно-респираторного синдрома свиней (РРСС); референс-штамм «Мозамбик-78» V генотипа вируса африканской чумы свиней (АЧС); изолят «Челябинск 2021» вируса диареи крупного рогатого скота II генотипа (ВД КРС), полевой изолят возбудителя рожи свиней Erysipelothrix rhusiopathiae. Патогенный биологический агент получен в форме лиофилизированного материала из государственной коллекции штаммов микроорганизмов и рабочей коллекции ФГБУ «Федеральный центр охраны здоровья животных» (ФГБУ «ВНИИЗЖ»).
Животные. Для получения первичной культуры клеток использовали подсвинков массой 10–15 кг в возрасте 2–2,5 мес. из благополучных по КЧС хозяйств Владимирской области. Эвтаназию и отбор эксплантата тестикул производили в соответствии с требованиями Директивы 2010/63/EU Европейского парламента и Совета Европейского союза от 22.09.2010 по охране животных, используемых в научных целях.
Культивирование. Вирус КЧС накапливали в первично трипсинизированной культуре клеток тестикул свиней (ТС), культивируемой на среде Игла-МЕМ, приготовленной по прописи ФГБУ «ВНИИЗЖ», с добавлением 10% фетальной сыворотки крови КРС и 50 мкг/см3 гентамицина сульфата [16]. Идентификацию репродукции возбудителя проводили в ОТ-ПЦР-РВ согласно методическим рекомендациям2.
Внутренний контрольный образец. В качестве ВКО выбран РНК-содержащий бактериофаг MS2 семейства Leviviridae, патогенный для Escherichia coli [17].
Дизайн праймеров и зондов. Выравнивание и сравнительный молекулярно-генетический анализ нуклеотидных последовательностей кДНК различных субгенотипов вируса КЧС, импортированных из базы данных GenBank, осуществляли с использованием программы Bioedit v7.2.5 и NCBI: Nucleotide BLAST. Критерием выбора оптимальных праймеров служили консервативные участки генома вируса КЧС. Праймеры и зонд для амплификации и гибридизации фрагмента ВКО выбраны исходя из литературных данных [18]. Синтез олигонуклеотидов выполнялся в компании ООО «Синтол» (Россия).
Выделение нуклеиновых кислот. РНК вируса КЧС экстрагировали из культуральной вируссодержащей суспензии ТС, а геном гетерологичных вирусов и бактерий – из лиофилизата нуклеосорбционным методом с использованием набора реагентов для выделения ДНК/РНК из биологического материала «РИБО-сорб» (ФБУН «Центральный НИИ эпидемиологии» Роспотребнадзора, Россия) в соответствии с инструкциями производителя3.
Постановка ОТ-ПЦР-РВ. Реакционная ПЦР-смесь содержала компоненты от производителя НПО «Евроген» (Россия), а именно набор OneTube RT-PCR TaqMan, включающий смесь OneTube RT-PCRmix, ревертазу TM-MMLV и воду, свободную от нуклеаз. Все стадии ПЦР (обратную транскрипцию, собственно амплификацию и гибридизационно-флуоресцентную детекцию) проводили в автоматическом амплификаторе Rotor-Gene Q в комплекте с программным обеспечением (QIAGEN, Германия).
Положительный контрольный образец (ПКО). В качестве ПКО предложен вакцинный штамм «СК» вируса КЧС с титром не менее 3,5 lg ККИД50/см3 в форме лиофилизированного материала, растворенного в 4,0 см3 физиологического раствора и инактивированного термическим методом путем нагревания в течение 60 мин при температуре +60 °C. Полноту инактивации проверяли методом трех последовательных слепых пассажей в культуре клеток ТС согласно методическим рекомендациям3.
Отрицательный контрольный образец (ОКО). В качестве ОКО использовали воду, свободную от нуклеаз, производства НПО «Евроген» (Россия).
Валидация. Валидационные параметры определялись согласно рекомендуемому руководству по публикации результатов разработки количественных ПЦР-РВ-протоколов (MIQE: Minimum Information for Publication of Quantitative Real-Time PCR Experiments) [19]. Для определения правильности, сходимости и воспроизводимости анализировали положительный на наличие вируса КЧС штамма «719» образец: в 6 повторяемых реакциях, в 3 параллельных испытаниях в течение дня одним исследователем и в 3 параллельных испытаниях в течение 3 дней двумя исследователями. Аналитическую чувствительность (предел детекции) вычисляли в 10-кратных разведениях вируса КЧС штаммов «719», «684», «Ши-мынь» с известным титром. Показатель предела детекции выражали в минимальном титре вируса (lg ККИД50/см3), обнаруженном валидируемым методом. Эффективность амплификации вычисляли согласно формуле:
Е = (101/slope − 1) × 100%,
где slope – это значение наклона линейной области зависимости Ct от логарифма концентрации кДНК-матрицы.
Проверку аналитической специфичности выполняли в реакции ОТ-ПЦР-РВ с заведомо отрицательными на наличие РНК вируса КЧС образцами, содержащими экстрагированный геном гетерологичных вирусов и бактерий, а также с образцами штаммов вируса КЧС различных субгенотипов. Для определения диагностической чувствительности готовили панель с 27 истинно положительными образцами (селезенка, печень, почка, кровь, лимфатические узлы, мышечная ткань, ректальные и оральные мазки), полученными на разных стадиях протекания инфекционного процесса у экспериментально зараженных эпизоотическим штаммом вируса КЧС свиней, а также образцами корма, контаминированного инфицированными животными. Показатель диагностической специфичности определяли путем исследования 27 заведомо отрицательных образцов полнорационного комбикорма для свиней, 10%-х суспензий селезенки, почек, печени, лимфатических узлов, мясных изделий свиного происхождения, цельной крови, оральных и ректальных смывов свободных от КЧС свиней, полученных в референтной лаборатории по африканской чуме свиней ФГБУ «ВНИИЗЖ» в рамках реализации государственного эпизоотологического мониторинга и проведения исследований на коммерческой основе со свиноводческими хозяйствами европейской части России. Сравнительная оценка как диагностической, так и аналитической чувствительности проводилась в параллельном исследовании образцов зашифрованными отечественными тест-системами № x1 и x2 согласно инструкции производителей. Выбранные тест-системы наиболее востребованы в России для диагностики КЧС и, так же как и исследуемая тест-система, содержат ВКО.
Статистическая обработка данных проводилась с использованием программы Microsoft Excel. Построение графиков выполнялось в программе GraphPad Prism.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Дизайн олигонуклеотидов. По результатам выравнивания и сравнительного анализа нуклеотидных последовательностей штаммов различных субгенотипов вируса КЧС наиболее консервативной областью генома, как и ожидалось, оказалась 5’-NTR, к которой и были подобраны прямой и обратный праймеры в модификации LNA, амплифицирующие фрагмент в 120 нуклеотидов (рис. 1).
TaqMan-зонды к целевому фрагменту генома вируса КЧС мечены флуорофором 6-FAM (6-карбоксифлуоресцеин), а к участку ВКО – флуорофором Cy5.5 (цианин-5.5).
Оптимизация условий ОТ-ПЦР-РВ. В процессе оптимизации определены температурно-временной профиль и количественный состав ПЦР-смеси.
Смесь на одну реакцию содержала 1Х OneTube RT-PCRmix, 0,4 пмоль прямого (forward) и 0,4 пмоль обратного (reverse) праймеров, 0,2 пмоль TaqMan-зонда, специфичных к фрагменту вируса КЧС; по 0,1 пмоль прямого, обратного праймеров и TaqMan-зонда, специфичных к участку ВКО, 1Х TM-MMLV ревертазы, 10,0 мкл РНК-матрицы. До конечного объема реакционной смеси 25,0 мкл добавлялась вода, свободная от нуклеаз.
Программа ПЦР включала в себя обратную транскрипцию при 50 °С в течение 25 мин и амплификацию с двойным циклированием: общую денатурацию с целью инактивации ревертазы и активации ДНК-полимеразы с «горячим стартом» при 95 °С в течение 10 мин, первые 10 циклов без детектирования флуоресценции (денатурация при 95 °С в течение 10 с, отжиг праймеров при 60 °С в течение 40 с, элонгация при 72 °С в течение 10 с), затем 35 циклов с детектированием флуоресценции на каналах Green и Crimson (денатурация при 95 °С в течение 10 с, отжиг праймеров при 55 °С в течение 40 с, элонгация при 72 °С в течение 10 с).
Выбор и оптимизация концентрации ВКО. В диагностике болезней, вызываемых РНК-содержащими вирусами (гепатит С, синдром приобретенного иммунодефицита человека и т. д.), в качестве ВКО используется бактериофаг МS2. Фаг семейства Leviviridae представляет собой небольшой вирион с икосаэдрическим типом симметрии, патогенный для Escherichia coli. Геном представлен 3569 нуклеотидами одноцепочечной РНК [20]. Основаниями для выбора MS2 как ВКО являются РНК-геном и безвредность для людей, животных, растений [21].
С целью определения достаточной концентрации ВКО для добавления в систему экстракции РНК проведено тестирование образцов интактной культуры клеток ТС, 10%-й суспензии селезенки зараженных эпизоотическим штаммом возбудителя и свободных от КЧС свиней, культурального вируса КЧС штамма «719» и ОКО с различным содержанием бактериофага MS2 в пробе (табл. 1).
В результате оптимальное количество ВКО составило 3,2 × 103 БОЕ бактериофага MS2 на 100 мкл пробы. В ходе оценки хранения суспензии MS2 с рекомендуемым титром при температуре +4 °С выяснили, что через шесть месяцев данный материал в ОТ-ПЦР-РВ имел стабильный пороговый цикл амплификации (Ct) с изменениями ± 2 Ct. При проведении пяти циклов заморозки материала при –20 °С и оттаивания при комнатной температуре Ct оставался стабильным.
Оценка правильности, сходимости и воспроизводимости. В результате исследования установлены 100%-е показатели правильности, сходимости и воспроизводимости валидируемого метода, так как заведомо положительный на наличие вируса КЧС штамма «719» образец в 6 повторяемых реакциях, в 3 параллельных испытаниях в течение дня одним исследователем и в 3 параллельных испытаниях в течение 3 дней двумя исследователями показал положительные результаты (табл. 2).
Однако, как обозначено на графике (рис. 2), на третий день испытания одного и того же образца значение Ct увеличивалось на 3,09 ± 0,81 в сравнении со вторым днем у обоих исследователей, что может быть связано с многократным замораживанием и оттаиванием вируссодержащего материала.
Определение аналитической чувствительности. Установлен средний показатель минимального титра вируса КЧС, детектируемого ОТ-ПЦР-РВ в 0,23 lg ККИД50/см3 для штаммов «719», «684» и «Ши-мынь».
Как представлено в таблице 3, сравнительный анализ параллельных исследований штаммов вируса разными тест-системами на основе ОТ-ПЦР-РВ показал, что предел детекции штамма «684» разработанным методом был на 1,2 lg выше, чем у тест-систем № х1 и х2, а предел детекции референтного штамма «Ши-мынь» был на 2,0 lg выше, чем у тест-системы № х2. При исследовании образца штамма «719» с титром 7,5 lg ККИД50/см3 валидируемым методом получены положительные результаты, в то время как параллельное испытание образца тест-системами № х1 и х2 приводило к сомнительным результатам.
С учетом всех полученных результатов Ct (n = 3) и 10-кратного разведения образца штамма вируса КЧС «719» построен график корреляции (рис. 3).
С помощью корреляционного анализа вычислены высокие статистические параметры, а именно: эффективность реакции Е = 105%, коэффициент адекватности R2 = 0,9928 и критерий значимости p value < 0,0001, что обуславливает перспективность использования ОТ-ПЦР-РВ в дальнейшей разработке количественной ПЦР со стандартными образцами.
Оценка аналитической специфичности. Образцы, содержащие геном гетерологичных вирусов и бактерий (возбудителей АЧС, болезни Ауески, РРСС, рожи свиней и ВД КРС), показали в ОТ-ПЦР-РВ отрицательный результат, а образцы, содержащие штаммы вируса КЧС, – положительный результат на наличие РНК вируса КЧС, соответственно аналитическая специфичность равна 100% (табл. 4).
Установлено, что при высоком показателе Ct на канале Green (геном вируса КЧС) возможна ингибиция амплификации фрагмента ВКО, что может быть связано с расходованием компонентов реакционной смеси (дезоксирибонуклеотидтрифосфатов, ДНК-полимеразы и др.) на синтез целевого ПЦР-продукта с большим числом копий матрицы [20]. Это обстоятельство необходимо учитывать при интерпретации результатов.
Определение диагностической чувствительности и специфичности. Все 27 истинно положительных образцов, содержащих вирус КЧС и полученных от экспериментально инфицированных эпизоотическим штаммом свиней, а также пробы контаминированного инфицированными животными корма в ОТ-ПЦР-РВ продемонстрировали положительный результат на наличие генома вируса КЧС. Аналогично все 27 истинно отрицательных проб показали отсутствие РНК вируса КЧС (рис. 4).
Таким образом, при проведении испытаний показатели диагностической чувствительности и специфичности представленного метода были максимально возможными и составили 100%. При этом положительный образец крови, полученный от экспериментально инфицированных эпизоотическим штаммом свиней, а также положительный образец корма, отобранный в виварном комплексе, где содержались экспериментально зараженные тем же штаммом свиньи, тест-системой № х2 на основе ОТ-ПЦР-РВ выявлены не были, при этом данная проба крови не была выявлена и тест-системой № х1, что указывает на более низкую диагностическую чувствительность (92,6% для тест-системы № х2 и 96,3% для тест-системы № х1), чем у валидируемого метода.
Анализ и интерпретация результатов. Рекомендуемые параметры ПЦР-анализа для каналов Green и Crimson тождественны: установка динамического фона, корректировки уклона, устранения выбросов на 10%, линейная шкала и пороговое значение, которое составляет 0,05. Результаты интерпретируются на основании наличия или отсутствия пересечения стандартной кривой с установленной пороговой линией, что соответствует наличию или отсутствию значения Ct в соответствующей графе таблицы результатов (рис. 5).
Также с целью установления параметров ПЦР-анализа и определения максимального Ct, при котором образец может интерпретироваться как положительный, проведено исследование двукратного разведения РНК, экстрагированной из суспензии вируса КЧС штамма «719» с итоговым титром 7,5 lg ККИД50/см3 (табл. 5).
Полученные данные показали, что максимальное значение Ct составило 27,83, что эквивалентно титру вируса КЧС штамма «719» 0,23 lg ККИД50/см3.
Результат считается достоверным, если получены правильные результаты для положительного и отрицательного контролей реакции.
Образец считается положительным на наличие генома вируса КЧС, если значение Ct на канале Green не превышает 28. Результат в таком случае является валидным вне зависимости показателей на канале Crimson.
Отрицательный результат на наличие генома вируса КЧС интерпретируют, если значение Ct на канале Green отсутствует, но на канале Crimson Ct не превышает 31.
Сомнительным является результат ПЦР при условии превышения 28 значения Ct на канале Green при значении Ct на канале Crimson менее 31.
При отсутствии значений Ct на каналах Green и Crimson, а также при превышении Ct более 31 на Crimson результат ПЦР является невалидным.
В случае сомнительного или невалидного результата необходимо провести повторное исследование пробы начиная с этапа выделения РНК с целью подтвердить или опровергнуть наличие генома вируса КЧС в данном образце.

Рис. 1. Выравнивание геномных последовательностей 5’-нетранслируемой области (5’-NTR) различных штаммов вируса КЧС, импортированных из GenBank (оранжевым цветом обозначены области отжига прямого и обратного праймеров, зеленым цветом – область гибридизации TaqMan-зонда)
Fig. 1. Alignment of CSF virus genome 5’-untranslated region (5’-NTR) sequences obtained from the GenBank (forward and reverse primer annealing sites are given in orange, TaqMan probe hybridization region is given in green)
Таблица 1
Результаты титрования ВКО в ОТ-ПЦР-РВ при использовании образцов различных материалов
Table 1
Results of internal control sample (ICS) titration with real-time RT-PCR using samples of different types
Образец | Титр ВКО (БОЕ/на реакцию) | Ct/ Green | Ct/ Crimson |
ОКО | 105 | – | 20,22 |
Интактная культура клеток ТС | – | 26,02 | |
10%-я суспензия селезенки свободной от КЧС свиньи | – | – | |
10%-я суспензия селезенки инфицированной вирусом КЧС свиньи | 14,28 | – | |
Вируссодержащая суспензия штамма «719» возбудителя КЧС | 6,15 | – | |
ОКО | 3,2 × 105 | – | 19,85 |
Интактная культура клеток ТС | – | 25,67 | |
10%-я суспензия селезенки свободной от КЧС свиньи | – | 22,80 | |
10%-я суспензия селезенки инфицированной вирусом КЧС свиньи | 13,62 | 23,22 | |
Вируссодержащая суспензия штамма «719» возбудителя КЧС | 5,66 | – | |
ОКО | 106 | – | 14,11 |
Интактная культура клеток ТС | – | 15,93 | |
10%-я суспензия селезенки свободной от КЧС свиньи | – | 17,90 | |
10%-я суспензия селезенки инфицированной вирусом КЧС свиньи | 15,12 | 14,73 | |
Вируссодержащая суспензия штамма «719» возбудителя КЧС | 5,95 | 26,07 | |
ОКО | 3,2 × 106 | – | 11,70 |
Интактная культура клеток ТС | – | 13,21 | |
10%-я суспензия селезенки свободной от КЧС свиньи | – | 15,21 | |
10%-я суспензия селезенки инфицированной вирусом КЧС свиньи | 12,10 | 10,48 | |
Вируссодержащая суспензия штамма «719» возбудителя КЧС | 5,56 | 24,25 | |
ОКО | 107 | – | 12,34 |
Интактная культура клеток ТС | – | 17,82 | |
10%-я суспензия селезенки свободной от КЧС свиньи | – | 11,93 | |
10%-я суспензия селезенки инфицированной вирусом КЧС свиньи | 14,43 | – | |
Вируссодержащая суспензия штамма «719» возбудителя КЧС | 8,37 | – | |
«–» – отрицательный результат (negative result); БОЕ – бляшкообразующая единица (plaque forming unit); Ct/Green – пороговое значение для детекции генома вируса КЧС (cycle threshold value for CSF virus genome detection); Ct/Crimson – пороговое значение для детекции ВКО (cycle threshold value for ICS detection). | |||
Таблица 2
Показатели правильности, сходимости и воспроизводимости метода ОТ-ПЦР-РВ
Table 2
Developed real-time RT-PCR assay accuracy, repeatability and reproducibility values
Правильность | ||||||
Образец | Ct/Green | (среднее значение ± SD) | Ct/Crimson | |||
Штамм «719» | 5,31 | 5,32 ± 0,148 | – | |||
Штамм «719» | 5,22 | 27,29 | ||||
Штамм «719» | 5,30 | 26,01 | ||||
Штамм «719» | 5,52 | 27,27 | ||||
Штамм «719» | 5,33 | 26,67 | ||||
Штамм «719» | 5,28 | 29,37 | ||||
Сходимость | ||||||
Порядок постановки реакции | Ct/Green | (среднее значение ± SD) | Ct/Crimson | |||
Первая постановка | 5,38 | 5,64 ± 0,237 | 28,63 | |||
Первая постановка | 5,74 | 26,85 | ||||
Первая постановка | 5,71 | 22,68 | ||||
Первая постановка | 5,61 | 23,78 | ||||
Первая постановка | 5,59 | 23,87 | ||||
Первая постановка | 5,86 | 24,41 | ||||
Вторая постановка | 5,63 | 5,44 ± 0,493 | 23,14 | |||
Вторая постановка | 5,37 | 23,95 | ||||
Вторая постановка | 5,02 | 23,62 | ||||
Вторая постановка | 5,11 | 23,11 | ||||
Вторая постановка | 5,96 | 23,01 | ||||
Вторая постановка | 5,56 | 23,70 | ||||
Третья постановка | 5,59 | 5,60 ± 0,095 | 28,84 | |||
Третья постановка | 5,60 | 30,30 | ||||
Третья постановка | 5,58 | 28,72 | ||||
Третья постановка | 5,74 | 30,26 | ||||
Третья постановка | 5,57 | 28,06 | ||||
Третья постановка | 5,57 | 28,87 | ||||
Воспроизводимость (Ct/Green) | ||||||
1-й день | 2-й день | 3-й день | ||||
первый исследователь | второй исследователь | первый исследователь | второй исследователь | первый исследователь | второй исследователь | |
5,52 | 5,07 | 5,67 | 6,51 | 7,83 | 8,95 | |
5,27 | 5,10 | 5,61 | 4,70 | 8,01 | 9,19 | |
5,46 | 5,82 | 5,65 | 4,55 | 6,71 | 9,17 | |
5,42 | 5,98 | 5,64 | 5,14 | 8,28 | 8,98 | |
5,38 | 5,33 | 5,73 | 5,44 | 8,02 | 9,30 | |
5,46 | 5,06 | 5,78 | 5,49 | 9,21 | 9,34 | |
5,4183 ± 0,125 | 5,3933 ± 0,591 | 5,68 ± 0,0917 | 5,305 ± 1,019 | 8,01 ± 1,166 | 9,155 ± 0,233 | |
«–» – отрицательный результат (negative result); Ct/Green – значение для фрагмента вируса КЧС (cycle threshold value for CSF virus fragment); Ct/Crimson – значение для фрагмента ВКО (cycle threshold value for ICS fragment). | ||||||
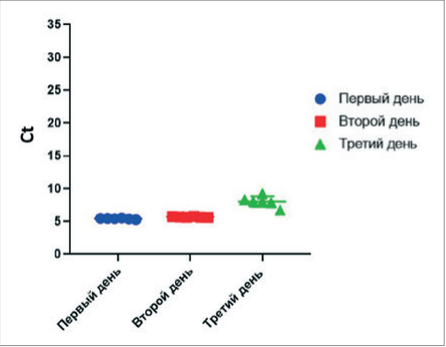
Рис. 2. Распределение показателей Ct для заведомо положительного образца в разные дни исследования при оценке воспроизводимости метода ОТ-ПЦР-РВ
Fig. 2. Distribution of Ct values for known positive sample in different days of testing when the real-time RT-PCR assay was assessed for its reproducibility
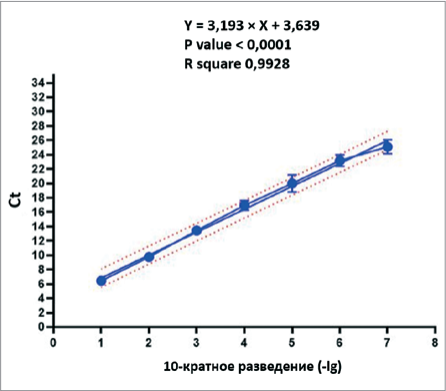
Рис. 3. График линейной корреляции разведений вируса КЧС штамма «719» с показателями Ct
Fig. 3. Graph of linear correlation of CSF virus “719” strain dilutions with Ct-values
Таблица 3
Результаты ОТ-ПЦР-РВ при 10-кратных разведениях культурального материала различных эпизоотических штаммов вируса КЧС
Table 3
Real-time RT-PCR results for 10-fold dilutions of various epizootic CSF virus strains
Разведение вируса | Штамм «Ши-мынь» (титр 4,0 lg ККИД50/см3) | Штамм «719» (титр 7,5 lg ККИД50/см3) | Штамм «684» (титр 6,2 lg ККИД50/см3) |
Ct/Green | Ct/Green | Ct/Green | |
Исходный материал | 11,88 (пол.) | 5,41 (пол.) | 6,79 (пол.) |
1:101 | 16,18 (пол.) | 9,75 (пол.) | 9,89 (пол.) |
1:102 | 20,52 (пол.) | 12,53 (пол.) | 13,69 (пол.) |
1:103 | 22,64 (пол.) | 16,68 (пол.) | 16,97 (пол.) |
1:104 | 27,06 (пол.) | 18,79 (пол.) | 20,24 (пол.) |
1:105 | – | 20,60 (пол.) | 23,45 (пол.) |
1:106 | – | 21,57 (пол.) | 26,30 (пол.) |
1:107 | – | 24,71 (пол.) | – |
1:108 | – | – | – |
Тест-система № х1 | |||
Ct/Yellow (результат) | |||
Исходный материал | 12,89 (пол.) | 6,44 (пол.) | 8,27 (пол.) |
1:101 | 16,77 (пол.) | 8,33 (пол.) | 12,89 (пол.) |
1:102 | 18,67 (пол.) | 13,56 (пол.) | 15,08 (пол.) |
1:103 | 21,96 (пол.) | 17,67 (пол.) | 17,36 (пол.) |
1:104 | 25,78 (пол.) | 18,82 (пол.) | 22,85 (пол.) |
1:105 | – | 24,01 (пол.) | 26,57 (сомнит.) |
1:106 | – | 22,79 (пол.) | – |
1:107 | – | 28,19 (сомнит.) | – |
1:108 | – | – | – |
Тест-система № х2 | |||
Ct/Yellow (результат) | |||
Исходный материал | 19,07 (пол.) | 10,60 (пол.) | 9,37 (пол.) |
1:101 | 22,42 (пол.) | 13,34 (пол.) | 12,69 (пол.) |
1:102 | 25,28 (пол.) | 16,46 (пол.) | 16,13 (пол.) |
1:103 | – | 17,67 (пол.) | 19,19 (пол.) |
1:104 | – | 20,55 (пол.) | 22,22 (пол.) |
1:105 | – | 22,55 (пол.) | 24,68 (пол.) |
1:106 | – | 25,44 (пол.) | – |
1:107 | – | 28,02 (сомнит.) | – |
1:108 | – | – | – |
«–» – отрицательный результат (negative result); пол. – положительный результат (positive result); сомнит. – сомнительный результат (inconclusive result); Ct/Green – значение для фрагмента вируса КЧС (cycle threshold value for CSF virus fragment); Ct/Yellow – значение для фрагмента вируса КЧС, полученное с использованием тест-систем № х1 и х2 на основе ПЦР-РВ согласно инструкции производителя (cycle threshold value for CSF virus fragment obtained when real-time PCR-based test systems No. х1 and х2 were used in accordance with their manufactures’ instructions). | |||
Таблица 4
Определение аналитической специфичности на наличие генома вируса КЧС в ОТ-ПЦР-РВ (n = 2)
Table 4
Assessment of analytical specificity of the real-time RT-PCR assay when the assay was used for CSF virus genome detection (n = 2)
Характеристика образца | Ct/Green | Ct/Crimson | Результат на наличие генома вируса КЧС |
Полевой изолят Erysipelothrix rhusiopathiae | – | 19,08 | отрицательный |
Штамм «Иркутский 2007» вируса РРСС | – | 13,03 | отрицательный |
Штамм «ВК»-ДЕП вируса болезни Ауески | – | 13,73 | отрицательный |
Штамм «Мозамбик-78» вируса АЧС | – | 15,39 | отрицательный |
Изолят «Челябинск 2021» возбудителя ВД КРС | – | 15,31 | отрицательный |
Референс-штамм вируса КЧС «Ши-мынь» | 18,64 | 15,39 | положительный |
Штамм вируса КЧС «CSF Amur 19-10/WB-12555» | 12,19 | 16,88 | положительный |
Штамм вируса КЧС «CSF Tigrovoe 16/WB-634» | 17,26 | 18,02 | положительный |
Штамм вируса КЧС «275» | 11,27 | – | положительный |
Штамм вируса КЧС «719» | 6,56 | – | положительный |
Штамм вируса КЧС «843» | 6,27 | – | положительный |
Штамм вируса КЧС «917» | 6,24 | – | положительный |
Штамм вируса КЧС «918» | 21,8 | 16,7 | положительный |
Штамм вируса КЧС «920» | 21,16 | 14,88 | положительный |
Штамм вируса КЧС «926» | 14,12 | 16,07 | положительный |
Штамм вируса КЧС «927» | 13,61 | 17,91 | положительный |
Штамм вируса КЧС «930» | 27,25 | 14,61 | положительный |
Штамм вируса КЧС «368» | 10,01 | 19,04 | положительный |
Штамм вируса КЧС «589» | 11,81 | 19,81 | положительный |
Штамм вируса КЧС «684» | 6,79 | 17,15 | положительный |
Штамм вируса КЧС «870» | 9,64 | 18,45 | положительный |
Штамм вируса КЧС «924» | 13,26 | 18,89 | положительный |
Штамм вируса КЧС «925» | 13,93 | 17,04 | положительный |
Штамм вируса КЧС «929» | 15,28 | 19,12 | положительный |
ОКО | – | 15,17 | отрицательный |
ПКО (вакцинный штамм вируса КЧС «СК») | 19,57 | 18,76 | положительный |
«–» – отрицательный результат (negative result); Ct/Green – значение для фрагмента вируса КЧС (cycle threshold value for CSF virus fragment); Ct/Crimson – значение для фрагмента ВКО (cycle threshold value for ICS fragment). | |||
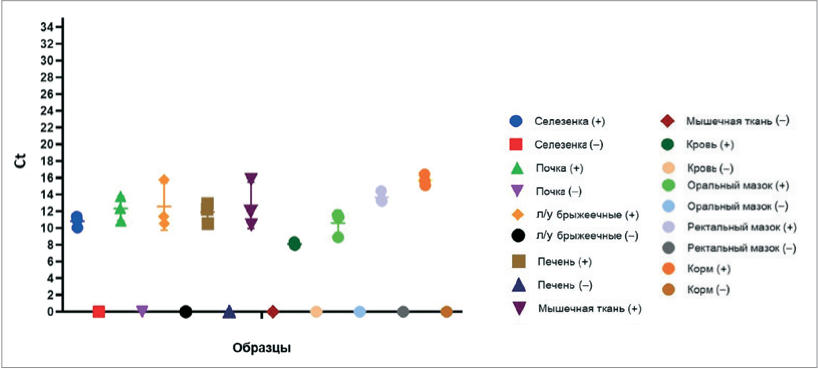
Рис. 4. Кинетика значений Ct для панели образцов, выбранной с целью определения диагностической чувствительности и специфичности (образцы – 10%-е суспензии указанных органов; «+» – истинно положительная проба; «–» – истинно отрицательная проба; л/у – лимфоузлы; в качестве отрицательных образцов мышечной ткани использованы мясное сырье и изделия свиного происхождения)
Fig. 4. Kinetics of Ct values for the panel of samples selected for determination of diagnostic sensitivity and specificity (samples – 10% suspensions of the indicated organs; «+» – true positive sample; «–» – true negative sample; l/n – lymph nodes; porcine raw meats and products were used as negative muscle samples)
Таблица 5
Результаты ОТ-ПЦР-РВ при двоичных разведениях РНК вируса КЧС
Table 5
Real-time RT-PCR results for two-fold dilutions of CSF virus RNA
Разведение РНК вируса КЧС | Ct/Green |
Исходный материал | 6,04 |
1:21 | 6,86 |
1:22 | 7,55 |
1:23 | 8,75 |
1:24 | 12,02 |
1:25 | 11,75 |
1:26 | 12,06 |
1:27 | 12,90 |
1:28 | 13,42 |
1:29 | 14,61 |
1:210 | 15,61 |
1:211 | 16,32 |
1:212 | 17,19 |
1:213 | 18,21 |
1:214 | 19,27 |
1:215 | 20,14 |
1:216 | 21,17 |
1:217 | 22,08 |
1:218 | 22,98 |
1:219 | 24,06 |
1:220 | 24,74 |
1:221 | 24,22 |
1:222 | 26,13 |
1:223 | 26,18 |
1:224 | 27,83 |
1:225 | 27,01 |
1:226 | – |
1:227 | – |
1:228 | – |
1:229 | – |
1:230 | – |
РНК вируса КЧС экстрагирована из вируссодержащей суспензии вируса КЧС штамма «719» c титром 7,5 lg ККИД50/см3 (CSF virus RNA was extracted from CSF virus “719” strain-containing suspension; titre 7.5 lg CCID50/cm3); «–» – отрицательный результат (negative result). | |
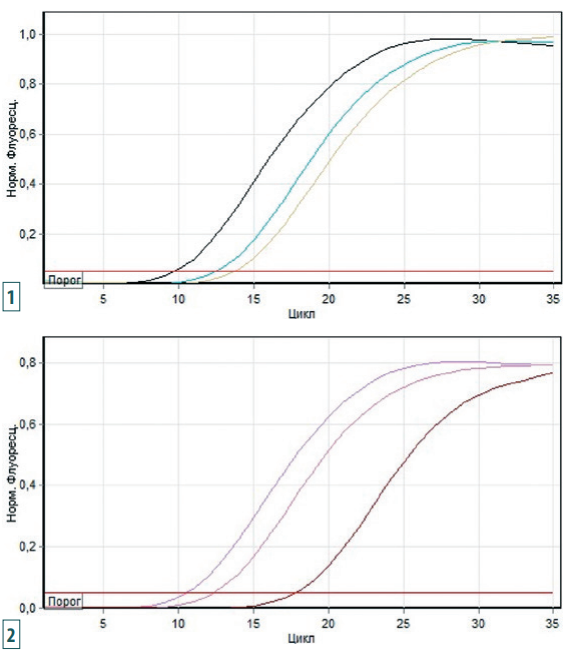
Рис. 5. Кривые флуоресценции: 1 – на канал Green (фрагмент вируса КЧС); 2 – на канал Crimson (фрагмент ВКО)
Fig. 5. Fluorescence curves: 1 – for Green channel (CSF virus fragment); 2 – for Crimson channel (ICS fragment)
ЗАКЛЮЧЕНИЕ
Разработанный метод обладает высокими валидационными характеристиками: 100%-ми показателями правильности, сходимости, воспроизводимости, аналитической специфичности, диагностической чувствительности и специфичности, пределом детекции 0,23 lg ККИД50/см3. Такие параметры делают предложенный метод ОТ-ПЦР-РВ конкурентоспособным на отечественном рынке диагностикумов и пригодным для использования в широкомасштабном мониторинге эпизоотической ситуации по КЧС в России.
1.
2. Колбин И. С., Власова Н. Н., Иголкин А. С., Елсукова А. А., Гаврилова В. Л., Пузанкова О. С. Методические рекомендации по выделению вируса классической чумы свиней на первичных культурах клеток (СС, КМС, СП, ТЯ, ТС) с идентификацией возбудителя методом полимеразной цепной реакции с гибридизационно-флуоресцентной детекцией в режиме реального времени: утв. ФГБУ «ВНИИЗЖ» 14.09.2021 № 42-21. Владимир: ФГБУ «ВНИИЗЖ»; 2021. 56 с.
3. Инструкция по применению комплекта реагентов для выделения РНК/ДНК из клинического материала «РИБО-сорб»: утв. приказом Росздравнадзора от 20.02.2009 № 1337-Пр/09. https://www.amplisens.ru/upload/iblock/259/RIBO-sorb.pdf
Список литературы
1. MalikY. S., Bhat S., KumarO. R. V., Yadav A. K., Sircar S., Ansari M. I., et al. Classical swine fever virus biology, clinicopathology, diagnosis, vaccines and a meta-analysis of prevalence: A review from the Indian perspective. Pathogens. 2020; 9 (6):500. https://doi.org/10.3390/pathogens9060500
2. Stark R., Meyers G., Rümenapf T., Thiel H. J. Processing of pestivirus polyprotein: cleavage site between autoprotease and nucleocapsid protein of classical swine fever virus. Journal of Virology. 1993; 67 (12): 7088–7095. https://doi.org/10.1128/jvi.67.12.7088-7095.1993
3. Blome S., Staubach C., Henke J., Carlson J., Beer M. Classical swine fever – an updated review. Viruses. 2017; 9 (4):86. https://doi.org/10.3390/v9040086
4. Edwards S., Fukusho A., Lefèvre P.-C., Lipowski A., Pejsak Z., Roehe P., Westergaard J. Classical swine fever: the global situation. Veterinary Microbiology. 2000; 73 (2–3): 103–119. https://doi.org/10.1016/s0378-1135(00)00138-3
5. Оганесян А. С., Шевцов А. А., Щербаков А. В., Коренной Ф. И., Караулов А. К. Классическая чума свиней: ретроспективный анализ эпизоотической ситуации в Российской Федерации (2007–2021 гг.) и прогноз на 2022 г. Ветеринария сегодня. 2022; 11 (3): 229–238. https://doi.org/10.29326/2304-196X-2022-11-3-229-238
6. Шевцов А. Классическая чума свиней: перспективы искоренения. Животноводство России. 2021; (10): 27–30. https://www.elibrary.ru/jfxalo
7. Zhu H., Zhang H., Xu Y., Laššáková S., Korabečná M., Neužil P. PCR past, present and future. BioTechniques. 2020; 69 (4): 317–325. https://doi.org/10.2144/btn-2020-0057
8. Hirschhorn J. W., Avery A., Schandl C. A. Managing a PCR contamination event in a molecular pathology laboratory. In: Clinical Applications of Nucleic Acid Amplification. Methodsin Molecular Biology. Eds. M. B. Myers, C. A. Schandl. 2023; 2621: 15–26. https://doi.org/10.1007/978-1-0716-2950-5_2
9. Canal C. W., Hotzel I., de Almeida L. L., Roehe P. M., Masuda A. Differentiation of classical swine fever virus from ruminant pestiviruses by reverse transcription and polymerase chain reaction (RT-PCR). Veterinary Microbiology. 1996; 48 (3–4): 373–379. https://doi.org/10.1016/0378-1135(95)00156-5
10. McGoldrick A., Lowings J. P., Ibata G., Sands J. J., Belak S., Paton D. J. A novel approach to the detection of classical swine fever virus by RT-PCR with a fluorogenic probe (TaqMan). Journal of Virological Methods. 1998; 72 (2): 125–135. https://doi.org/10.1016/s0166-0934(97)00208-5
11. Hoffmann B., Beer M., Schelp C., Schirrmeier H., Depner K. Validation of a real-time RT-PCR assay for sensitive and specific detection of classical swine fever. Journal of Virological Methods. 2005; 130 (1–2): 36–44. https://doi.org/10.1016/j.jviromet.2005.05.030
12. Latorra D., Arar K., Hurley J. M. Design considerations and effects of LNA in PCR primers. Molecular and Cellular Probes. 2003; 17 (5): 253–259. https://doi.org/10.1016/S0890-8508(03)00062-8
13. Monpoeho S., Coste-Burel M., Costa-Mattioli M., Besse B., Chomel J., Billaudel S., Ferre V. Application of a real-time polymerase chain reaction with internal positive control for detection and quantification of enterovirus in cerebrospinal fluid. European Journal of Clinical Microbiology and Infectious Diseases. 2002; 21 (7): 532–536. https://doi.org/10.1007/s10096-002-0766-5
14. Гаврилова В. Л., Пузанкова О. С., Латышев О. Е., Иголкин А. С., Манин Б. Л., Жбанова Т. В., Южаков А. Г. Изучение культуральных и молекулярных свойств изолятов вируса классической чумы свиней, выделенных в 2016 году. Труды Федерального центра охраны здоровья животных. 2017; 15: 82–101. https://www.elibrary.ru/xrdrrb
15. Гилеп А. А., Башко Н. П., Усанов С. А., Камышников В. С. Импортозамещающие и экспортоориентированные отечественные диагностические средства. Лабораторная диагностика. Восточная Европа. 2014; (4): 28–45. https://elibrary.ru/taxtgh
16. Колбин И. С., Иголкин А. С., Гаврилова В. Л., Пузанкова О. С., Аронова Е. В., Елсукова А. А., Власова Н. Н. Определение репродуктивных свойств вируса классической чумы свиней вирулентных и вакцинных штаммов в первичных и перевиваемых культурах клеток. Ветеринария сегодня. 2022; 11 (2): 149–155. https://doi.org/10.29326/2304-196X-2022-11-2-149-155
17. Dreier J., Störmer M., Kleesiek K. Use of bacteriophage MS2 as an internal control in viral reverse transcription-PCR assays. Journal of ClinicalMicrobiology. 2005; 43 (9): 4551–4557. https://doi.org/10.1128/jcm.43.9.4551-4557.2005
18. O’Connell K. P., Bucher J. R., Anderson P. E., Cao C. J., Khan A. S., Gostomski M. V., ValdesJ. J. Real-time fluorogenic reverse transcription-PCR assays for detection of bacteriophage MS2. Applied and Environmental Microbiology. 2006; 72 (1): 478–483. https://doi.org/10.1128/aem.72.1.478-483.2006
19. Bustin S. A., Benes V., Garson J. A., Hellemans J., Huggett J., Kubista M., et al. The MIQE guidelines: Minimum information for publication of quantitative real-time PCR experiments. Clinical Chemistry. 2009; 55 (4): 611–622. https://doi.org/10.1373/clinchem.2008.112797
20. Buckwalter S. P., Sloan L. M., Cunningham S. A., Espy M. J., Uhl J. R., Jones M. F., et al. Inhibition controlsfor qualitative real-time PCR assays: Are they necessary for all specimen matrices? Journal of Clinical Microbiology. 2014; 52 (6): 2139–2143. https://doi.org/10.1128/jcm.03389-13
21. Zambenedetti M. R., Pavoni D. P., Dallabona A. C., Dominguez A. C., Poersch C. de D., Fragoso S. P., Krieger M. A. Internal control for real-time polymerase chain reaction based on MS2 bacteriophage for RNA viruses diagnostics. Memórias do Instituto Oswaldo Cruz. 2017; 112 (5): 339–347. https://doi.org/10.1590/0074-02760160380
Об авторах
А. С. СадчиковаРоссия
Садчикова Анастасия Сергеевна - аспирант, ветеринарный врач референтной лаборатории по африканской чуме свиней
мкр. Юрьевец, г. Владимир, 600901
А. С. Иголкин
Россия
Иголкин Алексей Сергеевич - канд. вет. наук, заведующий референтной лабораторией по африканской чуме свиней
мкр. Юрьевец, г. Владимир, 600901
Р. С. Чернышев
Россия
Чернышев Роман Сергеевич - аспирант, ветеринарный врач референтной лаборатории по африканской чуме свиней
мкр. Юрьевец, г. Владимир, 600901
А. А. Козлов
Россия
Козлов Антон Александрович - канд. биол. наук, научный сотрудник референтной лаборатории вирусных болезней
мкр. Юрьевец, г. Владимир, 600901
И. С. Колбин
Россия
Колбин Иван Сергеевич - ветеринарный врач референтной лаборатории по африканской чуме свиней
мкр. Юрьевец, г. Владимир, 600901
А. В. Спрыгин
Россия
Спрыгин Александр Владимирович -д-р биол. наук, старший научный сотрудник референтной лаборатории болезней крупного рогатого скота, заведующий лабораторией молекулярных и генетических исследований
мкр. Юрьевец, г. Владимир, 600901
Д. А. Бирюченков
Россия
Бирюченков Дмитрий Анатольевич - канд. вет. наук, заведующий лабораторией профилактики болезней свиней
мкр. Юрьевец, г. Владимир, 600901
И. А. Чвала
Россия
Чвала Илья Александрович - канд. вет. наук, заместитель директора по НИР
мкр. Юрьевец, г. Владимир, 600901
А. Мазлум
Соединённые Штаты Америки
Мазлум Али - канд. биол. наук
г. Батон-Руж, LA 70803
Рецензия
Для цитирования:
Садчикова А.С., Иголкин А.С., Чернышев Р.С., Козлов А.А., Колбин И.С., Спрыгин А.В., Бирюченков Д.А., Чвала И.А., Мазлум А. Разработка и валидация высокочувствительного метода мультиплексной ОТ-ПЦР-РВ для обнаружения генома вируса классической чумы свиней. Ветеринария сегодня. 2024;13(3):223-233. https://doi.org/10.29326/2304-196X-2024-13-3-223-233
For citation:
Sadchikova A.S., Igolkin A.S., Chernyshev R.S., Kozlov A.A., Kolbin I.S., Sprygin A.V., Biryuchenkov D.A., Chvala I.A., Mazloum A. Development and validation of highly sensitive multiplex real-time RT-PCR assay for detection of classical swine fever virus genome. Veterinary Science Today. 2024;13(3):223-233. https://doi.org/10.29326/2304-196X-2024-13-3-223-233
JATS XML



































