Перейти к:
Разработка системы внутреннего контроля на основе штамма «Ла-Сота» вируса ньюкаслской болезни при диагностике бешенства методом ОТ-ПЦР
https://doi.org/10.29326/2304-196X-2025-14-3-249-254
Аннотация
Введение. На достоверность результатов, получаемых при проведении ПЦР-диагностики, могут влиять такие факторы, как ошибки оператора, неполадки в работе амплификатора, наличие в образце ингибиторов реакции, низкое качество реактивов и другое. Все это может приводить к появлению так называемых ложноотрицательных результатов.
Цель исследования. Разработка системы внутреннего контроля на основе гетерологичного вируса ньюкаслской болезни при детекции вируса бешенства методом полимеразной цепной реакции.
Материалы и методы. В качестве внутреннего контрольного образца использовалась «Вакцина против ньюкаслской болезни из штамма «Ла-Сота» сухая живая» производства ФГБУ «ВНИИЗЖ» (Россия). РНК из образцов выделяли с помощью набора реагентов «РИБО-сорб» (ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора, Россия). Для полимеразной цепной реакции с обратной транскрипцией использовали реактивы фирмы Promega (США) и олигонуклеотиды производства ООО «Синтол» (Россия).
Результаты. В качестве объекта для внутреннего контрольного образца выбран штамм «Ла-Сота» вируса ньюкаслской болезни. Проведен дизайн праймеров. В серии экспериментов установлено, что ПЦР-система для внутреннего контрольного образца не конкурирует с ПЦР-системой для вируса бешенства при их совместном использовании. Оптимизированы основные параметры обратной транскрипции и полимеразной реакции. Проведена валидация разработанной методики, в ходе которой определялись такие характеристики, как правильность, специфичность, чувствительность, промежуточная прецизионность в условиях повторяемости (сходимость) и промежуточная прецизионность в условиях воспроизводимости (воспроизводимость). По результатам валидации полученные характеристики метода соответствуют требуемым.
Заключение. На основе штамма «Ла-Сота» вируса ньюкаслской болезни разработана система внутреннего контрольного образца для использования совместно с методикой выявления вируса бешенства методом полимеразной цепной реакции с обратной транскрипцией, позволяющая контролировать ход всех этапов анализа в каждой реакционной пробирке. Данная система при надлежащей оптимизации потенциально может применяться также в экспериментальных научных исследованиях в соответствующих профильных научных организациях при ПЦР-диагностике заболеваний, вызываемых другими РНК-содержащими вирусами.
Ключевые слова
Для цитирования:
Чупин С.А. Разработка системы внутреннего контроля на основе штамма «Ла-Сота» вируса ньюкаслской болезни при диагностике бешенства методом ОТ-ПЦР. Ветеринария сегодня. 2025;14(3):249-254. https://doi.org/10.29326/2304-196X-2025-14-3-249-254
For citation:
Chupin S.A. Construction of Newcastle disease virus LaSota strain-based internal sample for rabies diagnosis with RT-PCR. Veterinary Science Today. 2025;14(3):249-254. https://doi.org/10.29326/2304-196X-2025-14-3-249-254
ВВЕДЕНИЕ
На достоверность результатов, получаемых при проведении ПЦР-диагностики, могут влиять такие факторы, как ошибки оператора, неполадки в работе амплификатора, наличие в образце ингибиторов полимеразной цепной реакции (ПЦР), низкое качество реактивов и другое [1]. Все это может приводить к появлению так называемых ложноотрицательных результатов, когда в ходе анализа получают отрицательный результат, несмотря на то что искомый агент содержится в анализируемом образце. Одним из самых действенных методов, направленных на то, чтобы сделать ПЦР-анализ более достоверным, является использование внутренних контрольных образцов (ВКО) [2]. ВКО – это определенный объект, содержащий нуклеиновую кислоту, который добавляется к тестируемому образцу и проходит вместе с ним все или некоторые этапы анализа. При этом происходит специфическая амплификация фрагмента нуклеиновой кислоты этого объекта, по наличию которой можно судить о надлежащем ходе анализа.
К настоящему времени в литературе описано достаточное количество методов обнаружения генома вируса бешенства на основе ПЦР, как в ее классическом варианте [3-9], так и в варианте «в режиме реального времени» [10-18]. Однако использование ВКО включают только некоторые из них.
В целом ВКО широко применяется при ПЦР-диагностике различных патогенов. При этом существуют различные стратегии дизайна ВКО. Так, J. Coertse et al. использовали искусственно синтезированную РНК, последовательность которой соответствовала фрагменту генома вируса бешенства штамма CVS [19]. Smith J. et al. в качестве внутреннего контроля использовали рибосомальную РНК, которая, по их утверждению, имеет кинетику деградации, сходную с вирусной РНК [20]. В ряде работ описано применение ВКО из бактериофага MS2 [21-24]. Наряду с этим использовались также полученные генно-инженерным способом вирусоподобные частицы [25-27]. Одним из классических объектов для ВКО являются плазмиды, содержащие вставку с участками, отвечающими праймерам для целевой ПЦР [28-31]. Также описан метод использования ВКО на основе гетерологичных вирусов [32][33].
На протяжении ряда лет в референтной лаборатории по бешенству и BSE ФГБУ «ВНИИЗЖ» (г. Владимир) для обнаружения вируса бешенства наряду с прочими методами используется метод на основе ПЦР, разработанный и описанный A. E. Metlin et al. [34], обладающий высокой чувствительностью и специфичностью и дающий стабильные результаты. Современные стандарты, однако, накладывают требования увеличения степени достоверности результатов. Поэтому в целях повышения качества исследований была поставлена цель разработки и внедрения в ПЦР-диагностику бешенства использования ВКО.
МАТЕРИАЛЫ И МЕТОДЫ
Материалом служили образцы головного мозга животных, поступающие на исследование на наличие вируса бешенства в референтную лабораторию по бешенству и BSE ФГБУ «ВНИИЗЖ» (г. Владимир).
Для оптимизации полимеразной цепной реакции с обратной транскрипцией (ОТ-ПЦР) и в качестве положительного контрольного образца использовался штамм вируса бешенства «ВНИИЗЖ» из коллекции штаммов микроорганизмов ФГБУ «ВНИИЗЖ» с инфекционной активностью в культуре клеток 6,0 lgТЦД50/см3.
В качестве ВКО применялась «Вакцина против ньюкаслской болезни из штамма «Ла-Сота» сухая живая», фасовка 4000 доз, содержание вируса ньюкаслской болезни – 4 × 109,7 ЭИД50 (ФГБУ «ВНИИЗЖ», г. Владимир).
РНК из образцов выделяли с помощью набора реагентов «РИБО-сорб» (ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора, Россия) согласно инструкции производителя.
ОТ-ПЦР осуществляли, как описано A. E. Metlin et al. [34].
Для дизайна праймеров использовали программу Amplicon (версия b08) [35] и последовательность штамма вируса ньюкаслской болезни «Ла-Сота» (номер доступа в базе данных GenBank – JF950510).
Для постановки ОТ-ПЦР применяли реагенты фирмы Promega (США).
Праймеры были синтезированы в ООО «Синтол» (Россия).
Анализ продуктов ПЦР осуществляли с помощью электрофореза в 2%-м агарозном геле, содержащем бромистый этидий. Для фиксации электрофореграммы использовали гель-документирующую систему GelDoc (Bio-Rad Laboratories, Inc., США).
Полученные ампликоны секвенировали с применением праймеров, которые использовались в ОТ-ПЦР, и набора BigDye Terminator Cycle Sequencing kit (Applied Biosystems, США) на капиллярном ДНК-секвенаторе ABI Prism 3100 (Applied Biosystems, США).
Для проведения валидации в качестве положительного стандартного контрольного образца был использован штамм РВ-97 вируса бешенства (получен из коллекции штаммов микроорганизмов ФГБУ «ВНИИЗЖ»), в качестве отрицательного стандартного контрольного образца – дистиллированная вода. Положительными контрольными образцами служили пробы головного мозга различных животных, наличие вируса бешенства в которых было подтверждено в реакции иммунофлюоресценции (РИФ). В качестве отрицательных контрольных образцов были использованы образцы головного мозга различных животных, отсутствие вируса бешенства в которых было подтверждено методом РИФ.
Правильность методики оценивали тестированием положительного стандартного контрольного образца в 10 повторностях и отрицательного стандартного контрольного образца в 10 повторностях.
Для определения специфичности исследовали 10 проб головного мозга, отсутствие вируса бешенства в которых было подтверждено методом РИФ. Специфичность рассчитывали как процентное отношение истинно отрицательных результатов к общему количеству исследований по формуле:
Sp = (ИО / (ИО + ЛП)) × 100%,
где ИО – истинно отрицательный результат;
ЛП – ложноположительный результат.
Для определения чувствительности исследовали 10 проб головного мозга, наличие вируса бешенства в которых было подтверждено методом РИФ. Чувствительность рассчитывали как процентное отношение истинно положительных результатов к общему количеству исследований по формуле:
Se = (ИП / (ИП + ЛО)) × 100%,
где ИП – истинно положительный результат;
ЛО – ложноотрицательный результат.
При определении промежуточной прецизионности в условиях повторяемости (сходимость) исследовали одну положительную пробу в трех повторностях, выполненных в одних и тех же условиях измерения (оборудование, оператор, лаборатория и короткий промежуток времени, то есть в условиях повторяемости).
При определении промежуточной прецизионности в условиях воспроизводимости (воспроизводимость) исследовали одну положительную пробу в трех повторностях, выполненных при измененных условиях измерения: одним исследователем в параллельных исследованиях в течение разных дней (три дня) и двумя разными исследователями в параллельных исследованиях (в трех повторах) в течение одного дня.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Одним из наиболее оптимальных типов системы ВКО при детекции вирусов методами молекулярной диагностики является применение гетерологичных вирусов с аналогичной структурой генома (ДНК/РНК, одна/две нити). Эта стратегия позволяет проконтролировать все стадии анализа начиная от выделения нуклеиновой кислоты, продолжая постановкой обратной транскрипции и заканчивая проведением ПЦР. При этом контрольный объект имеет максимально схожую с исследуемым природу и проходит через все стадии анализа в одном и том же реакционном сосуде (пробирке).
В качестве объекта для ВКО был выбран штамм «Ла-Сота» вируса ньюкаслской болезни, входящий в состав сухой живой вакцины против этой болезни. Основанием для такого выбора послужило то, что, будучи представителем семейства Paramyxoviridae, возбудитель ньюкаслской болезни, подобно вирусу бешенства, обладает геномом из однонитевой РНК негативной полярности. Кроме того, вакцина является фактически готовым к применению препаратом ВКО и требует только разбавления водой, поэтому выпадают такие трудоемкие этапы, как культивирование и титрование вируса.
Был проведен дизайн шести различных вариантов пар праймеров. Праймеры соответствовали области генов, кодирующих нуклеопротеин, фосфопротеин и матриксный протеин, а также 3’-некодирующей области генома штамма «Ла-Сота» вируса ньюкаслской болезни. При дизайне праймеров учитывалась температура отжига, при которой проходит целевая реакция (55 °C). Расчетная длина фрагмента, получаемого при амплификации ВКО (около 700 п. н. для всех пар), была выбрана заведомо больше, чем длина целевого фрагмента (384 п. н.), с тем чтобы в ходе ПЦР наработка целевого фрагмента имела конкурентные преимущества перед наработкой фрагмента ВКО [36]. Специфичность дизайна праймеров была проверена с помощью интернет-сервиса Primer-BLAST (www.ncbi.nlm.nih.gov/tools/primer-blast).
Проверка работоспособности синтезированных пар праймеров проводилась с использованием вакцины против ньюкаслской болезни из штамма вируса «Ла-Сота» (рис. 1).

Рис. 1. Электрофореграмма продуктов ПЦР с различными комбинациями праймеров для амплификации фрагмента генома штамма «Ла-Сота» вируса ньюкаслской болезни. Каждая комбинация представлена в двух повторностях. Цифрами обозначены дорожки с комбинациями праймеров: 1 – LASF815 – LASR1500; 2 – LASF1632 – LASR2337; 3 – LASF2086 – LASR2793; 4 – LASF2318 – LASR3026; 5 – LASF2598 – LASR3296; 6 – LASF107 – LASR800. М – маркер молекулярных весов GeneRuler 100 bp Plus DNA Ladder (ThermoFisher Scientific) с шагом длины фрагментов 100 п. н.
Fig. 1. Electropherogram of the PCR products using different combinations of primers for amplification Newcastle disease virus LaSota strain genome fragment. Each combination is given in duplicate. The numbers indicate the lanes with the following primer combinations: 1 – LASF815 – LASR1500; 2 – LASF1632 – LASR2337; 3 – LASF2086 – LASR2793; 4 – LASF2318 – LASR3026; 5 – LASF2598 – LASR3296; 6 – LASF107 – LASR800. М – molecular weight marker GeneRuler 100 bp Plus DNA Ladder (Thermo Fisher Scientific) fragment length is 100 bp
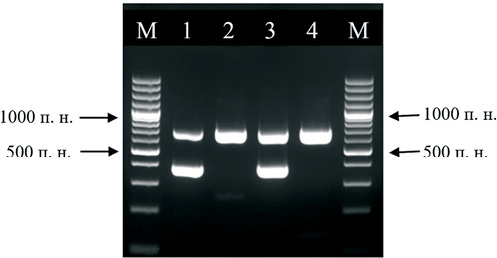
Рис. 2. Электрофореграмма продуктов ПЦР с образцами, содержащими и не содержащими вирус бешенства. Цифрами 1 и 3 обозначены дорожки с образцами, содержащими вирус бешенства; цифрами 2 и 4 – не содержащими вирус бешенства. М – маркер молекулярных весов GeneRuler 100 bp Plus DNA Ladder (ThermoFisher Scientific) с шагом длины фрагментов 100 п. н.
Fig. 2. Electropherogram of PCR products from rabies virus-positive samples and rabies virus-negative samples. The numbers 1 and 3 indicate the lanes with rabies virus-positive samples; the numbers 2 and 4 indicate the lanes with rabies virus-negative samples. М – molecular weight marker GeneRuler 100 bp Plus DNA Ladder (Thermo Fisher Scientific) fragments length is 100 bp
По результатам проверки были выбраны три лучшие комбинации праймеров: LASF107 – LASR800, LASF2086 – LASR2793, LASF2318 – LASR3026. Каждая из этих пар была испытана вместе с целевой мишенью, чтобы определить, как две системы влияют друг на друга. Было показано, что применение двух систем в комбинации в каждом из трех вариантов не дает нежелательных эффектов. Кроме того, с помощью серии десятикратных разведений вакцины была проведена оценка аналитической чувствительности выявления ВКО с помощью выбранных пар праймеров. Наибольшую чувствительность (разведение исходной вакцины 1:10000) показала пара праймеров LASF107 – LASR800, поэтому она была окончательно выбрана для системы ВКО. Данная пара праймеров позволяет амплифицировать фрагмент генома вируса ньюкаслской болезни размером 693 нуклеотида. Специфичность получаемого в ходе амплификации фрагмента ВКО была подтверждена путем нуклеотидного секвенирования.
В серии экспериментов было установлено, что с увеличением концентрации вируса бешенства чувствительность системы обнаружения ВКО падает; с другой стороны, с увеличением концентрации ВКО чувствительность системы обнаружения вируса бешенства остается прежней. Таким образом, система праймеров для выявления ВКО не конкурирует с системой праймеров для выявления вируса бешенства, что важно при проведении экспертизы в случае, когда в материале присутствует малое количество вируса бешенства. Однако для увеличения степени надежности целевой ПЦР для ВКО была выбрана рабочая концентрация, лишь немного превосходящая минимальную, а именно 1:1000 от исходной вакцины. Пример работы двух систем праймеров в присутствии ВКО приведен на рисунке 2.
Была проведена серия экспериментов по оптимизации основных параметров обратной транскрипции и полимеразной реакции. Установлено, что оптимальными параметрами ПЦР являются: концентрация ионов магния – 3 мМ, концентрация праймеров – 40 мМ. ВКО в виде вакцины, разбавленной водой в 1000 раз, в количестве 10 мкл добавляется в испытуемый образец (50 мкл суспензии головного мозга или 100 мкл культуральной жидкости) на этапе выделения РНК.
Таким образом, была получена методика, позволяющая проводить детекцию вируса бешенства в образцах и одновременно контролировать качество анализа в каждой реакционной пробирке. При проведении анализа наряду с образцами тестируются также заведомо положительный и заведомо отрицательный образцы (содержащие и не содержащие вирус бешенства – внешние положительный и отрицательный контроль), в которые также добавляют по 10 мкл ВКО. Наличие в продуктах ПЦР фрагмента ДНК размером 693 п. н. при отсутствии фрагмента размером 384 п. н. свидетельствует о корректном проведении анализа.
С помощью полученной методики было протестировано 52 образца, положительных на наличие вируса бешенства в РИФ, и 48 отрицательных образцов. Во всех случаях результаты детекции вируса бешенства совпали с результатами РИФ. При тестировании отрицательных образцов в продуктах ПЦР всегда присутствовал фрагмент размером 693 п. н., что свидетельствовало об амплификации фрагмента генома ВКО, то есть об отсутствии ложноположительного результата. При анализе положительных на наличие вируса бешенства образцов фрагмент размером 693 п. н. амплифицировался не всегда, что является абсолютно корректным результатом, так как целевая реакция имеет конкурентное преимущество перед реакцией с ВКО – все ресурсы в реакционной пробирке расходуются на синтез целевого фрагмента. В то же время при анализе положительных на вирус бешенства образцов в продуктах реакции всегда присутствовал фрагмент размером 384 п. н., свидетельствующий о наличии в образце вируса бешенства.
С целью установления надежности разработанной методики была проведена ее валидация, в ходе которой оценивались такие характеристики, как правильность, специфичность, чувствительность, промежуточная прецизионность в условиях повторяемости (сходимость) и промежуточная прецизионность в условиях воспроизводимости (воспроизводимость). При определении правильности для всех проб был получен результат, совпадающий со статусом пробы. Таким образом, рассчитанная правильность валидируемого метода составила 100%. При установлении специфичности все исследованные пробы с отсутствием вируса бешенства показали в валидируемом методе отрицательный результат. Следовательно, рассчитанная специфичность валидируемого метода составила 100%. При определении чувствительности все исследованные пробы с наличием вируса бешенства показали в валидируемом методе положительный результат, из чего следует, что рассчитанная чувствительность валидируемого метода составила 100%. При оценке сходимости во всех трех повторностях был получен положительный результат. Таким образом, было показано, что сходимость валидируемого метода абсолютна. При определении воспроизводимости во всех случаях были получены положительные результаты. Следовательно, для валидируемого метода воспроизводимость была абсолютна. По результатам валидации полученные характеристики метода соответствуют требуемым.
При всем разнообразии применяемых для ВКО дизайнов каждый из них имеет свои преимущества и недостатки. Так, искусственно синтезированная РНК [19] подвержена разрушительному действию РНК-рестриктаз, которые в норме в изобилии присутствуют в образце. Рибосомальная РНК, использованная J. Smith et al. [20], за счет развитых третичных структур в принципе более устойчива к действию рестриктаз, но насколько надежно и в каком спектре условий – еще предстоит выяснить. Бактериофаг MS2, будучи вирусной частицей, лишен этого недостатка, так как обладает белковой оболочкой, защищающей его от рестриктаз. Однако культивирование фага представляет собой отдельный этап работы. Искусственно созданные вирусоподобные частицы имеют много преимуществ, включая наличие белковой оболочки, нуклеотидной последовательности генома с заданными свойствами, нужным типом нуклеиновой кислоты. В то же время создание таких частиц высокотехнологично, а поддержание трудоемко. Изготовление плазмид, которые используются в качестве ВКО во многих отечественных коммерческих диагностикумах, при налаженном производстве не требует больших экономических затрат. Однако такая стратегия ограничивает тип нуклеиновой кислоты одной лишь ДНК, а в случае использования при диагностике РНК-вирусов исключается контроль такого важного этапа, как обратная транскрипция. Одной из наиболее удачных, на наш взгляд, стратегий дизайна ВКО сочетающей в себе многие преимущества других подходов, является использование гетерологичных вирусов. Во-первых, они обладают надежной белковой оболочкой, защищающей их от действия рестриктаз. Во-вторых, можно выбрать вирус с типом нуклеиновой кислоты, как у целевого вируса (ДНК/РНК, одна/две нити). В-третьих, это уже готовые биологические объекты, и их можно сравнительно легко размножать в культуре клеток. При использовании же готового препарата в виде вакцины (как в нашем случае) исключаются даже этапы культивирования и титрования в культуре клеток.
Стоит отметить, что разработанная система ВКО на основе штамма «Ла-Сота» вируса ньюкаслской болезни может также применяться в имеющемся виде или после доработки (в случае несовпадения температурно-временных параметров ПЦР с целевой системой) и при ПЦР-диагностике заболеваний, вызываемых другими РНК-содержащими вирусами.
ЗАКЛЮЧЕНИЕ
На основе штамма «Ла-Сота» вируса ньюкаслской болезни разработана система ВКО для использования совместно с методикой выявления вируса бешенства методом ОТ-ПЦР, позволяющая контролировать ход всех этапов анализа в каждой реакционной пробирке. Надежность работы данной системы проверена на примере клинических образцов, содержащих и не содержащих вирус бешенства. Была проведена успешная валидация разработанной системы. Данная система, при надлежащей оптимизации, потенциально может применяться также в экспериментальных научных исследованиях в соответствующих профильных научных организациях при ПЦР-диагностике заболеваний, вызываемых другими РНК-содержащими вирусами.
Список литературы
1. Hoorfar J., Malorny B., Abdulmawjood A., Cook N., Wagner M., Fach P. Practical considerations in design of internal amplification controls for diagnostic PCR assays. Journal of Clinical Microbiology. 2004; 42 (5): 1863–1868. https://doi.org/10.1128/JCM.42.5.1863-1868.2004
2. Buckwalter S. P., Sloan L. M., Cunningham S. A., Espy M. J., Uhl J. R., Jones M. F., et al. Inhibition controls for qualitative real-time PCR assays: are they necessary for all specimen matrices? Journal of Clinical Microbiology. 2014; 52 (6): 2139–2143. https://doi.org/10.1128/JCM.03389-13
3. Favoretto S. R., Martorelli L. F. А., Elkhoury M. R., Zargo A. M., Durigon E. L. Rabies virus detection and phylogenetic studies in samples from an exhumed human. Clinical Infectious Diseases. 2005; 41 (3): 413–414. https://doi.org/10.1086/431766
4. Smith J. S., Orciari L. A., Yager P. A. Molecular epidemiology of rabies in the United States. Seminars in Virology. 1995; 6 (6): 387–400. https://doi.org/10.1016/S1044-5773(05)80016-2
5. De Mattos C. C., De Mattos C. A., Loza-Rubio E., Aguilar-Setién A., Orciari L. A., Smith J. S. Molecular characterization of rabies virus isolates from Mexico: implications for transmission dynamics and human risk. American Journal of Tropical Medicine and Hygiene. 1999; 61 (4): 587–597. https://doi.org/10.4269/ajtmh.1999.61.587
6. Dacheux L., Reynes J. M., Buchy P., Sivuth O., Diop B. M., Rousset D., et al. A reliable diagnosis of human rabies based on analysis of skin biopsy specimens. Clinical Infectious Diseases. 2008; 47 (11): 1410–1417. https://doi.org/10.1086/592969
7. Araújo D. B., Langoni H., Almeida M. F., Megid J. Heminested reverse-transcriptase polymerase chain reaction (hnRT-PCR) as a tool for rabies virus detection in stored and decomposed samples. BMC Research Notes. 2008; 1:17. https://doi.org/10.1186/1756-0500-1-17
8. David D., Yakobson B., Rotenberg D., Dveres N., Davidson I., Stram Y. Rabies virus detection by RT-PCR in decomposed naturally infected brains. Veterinary Microbiology. 2002; 87 (2): 111–118. https://doi.org/10.1016/s0378-1135(02)00041-x
9. Heaton P. R., Johnstone P., McElhinney L. M., Cowley R., O’Sullivan E., Whitby J. E. Heminested PCR assay for detection of six genotypes of rabies and rabies-related viruses. Journal of Clinical Microbiology. 1997; 35 (11): 2762–2766. https://doi.org/10.1128/jcm.35.11.2762-2766.1997
10. Nadin-Davis S. A., Sheen M., Wandeler A. I. Development of real-time reverse transcriptase polymerase chain reaction methods for human rabies diagnosis. Journal of Medical Virology. 2009; 81 (8): 1484–1497. https://doi.org/10.1002/jmv.21547
11. Wacharapluesadee S., Sutipanya J., Damrongwatanapokin S., Phumesin P., Chamnanpood P., Leowijuk C., Hemachudha T. Development of a TaqMan real-time RT-PCR assay for the detection of rabies virus. Journal of Virological Methods. 2008; 151 (2): 317–320. https://doi.org/10.1016/j.jviromet.2008.05.004
12. Landry M. L., Garner R., Ferguson D. Real-time nucleic acid sequence-based amplification using molecular beacons for detection of enterovirus RNA in clinical specimens. Journal of Clinical Microbiology. 2005; 43 (7): 3136–3139. https://doi.org/10.1128/JCM.43.7.3136-3139.2005
13. Hayman D. T. S., Banyard A. C., Wakeley P. R., Harkess G., Marston D., Wood J. L. N., et al. A universal real-time assay for the detection of Lyssaviruses. Journal of Virological Method. 2011; 177 (1): 87–93. https://doi.org/10.1016/j.jviromet.2011.07.002
14. Wakeley P. R., Johnson N., McElhinney L. M., Marston D., Sawyer J., Fooks A. R. Development of a real-time, TaqMan reverse transcription-PCR assay for detection and differentiation of lyssavirus genotypes 1, 5, and 6. Journal of Clinical Microbiology. 2005; 43 (6): 2786–2792. https://doi.org/10.1128/JCM.43.6.2786-2792.2005
15. Hoffmann B., Freuling C. M., Wakeley P. R., Rasmussen T. B., Leech S., Fooks A. R., et al. Improved safety for molecular diagnosis of classical rabies viruses by use of a TaqMan real-time reverse transcription-PCR“double check” strategy. Journal of Clinical Microbiology. 2010; 48 (11): 3970–3978. https://doi.org/10.1128/JCM.00612-10
16. Wadhwa A., Wilkins K., Gao J., Condori Condori R. E., Gigante C. M., Zhao H., et al. A Pan-Lyssavirus Taqman Real-Time RT-PCR Assay for the detection of highly variable Rabies virus and other lyssaviruses. PLoSNeglected Tropical Diseases. 2017; 11 (1):e0005258. https://doi.org/10.1371/journal.pntd.0005258
17. Dacheux L., Larrous F., Lavenir R., Lepelletier A., Faouzi A., Troupin C., et al. Dual combined real-time reverse transcription polymerase chain reaction assay for the diagnosis of lyssavirus infection. PLoS Neglected Tropical Diseases. 2016; 10 (7):e0004812. https://doi.org/10.1371/journal.pntd.0004812
18. Suin V., Nazé F., Francart A., Lamoral S., De Craeye S., Kalai M., Van Gucht S. A two-step lyssavirus real-time polymerase chain reaction using degenerate primers with superior sensitivity to the fluorescent antigen test. BioMed Research International. 2014; 2014:256175. https://doi.org/10.1155/2014/256175
19. Coertse J., Weyer J., Nel L. H., Markotter W. Improved PCR methods for detection of African rabies and rabies-related lyssaviruses. Journal of Clinical Microbiology. 2010; 48 (11): 3949–3955. https://doi.org/10.1128/JCM.01256-10
20. Smith J., McElhinney L. M., Heaton P. R., Black E. M., Lowings J. P. Assessment of template quality by the incorporation of an internal control into a RT-PCR for the detection of rabies and rabies-related viruses. Journal of Virological Methods. 2000; 84 (2): 107–115. https://doi.org/10.1016/s0166-0934(99)00124-x
21. Zambenedetti M. R., Pavoni D. P., Dallabona A. C., Dominguez A. C., Poersch C. O., Fragoso S. P., Krieger M. A. Internal control for real-time polymerase chain reaction based on MS2 bacteriophage for RNA viruses diagnostics. Memórias do Instituto Oswaldo Cruz. 2017; 112 (5): 339–347. https://doi.org/10.1590/0074-02760160380
22. Dreier J., Störmer M., Kleesiek K. Use of bacteriophage MS2 as an internal control in viral reverse transcription-PCR assays. Journal of Clinical Microbiology. 2005; 43 (9): 4551–4557. https://doi.org/10.1128/JCM.43.9.4551-4557.2005
23. Felder E., Wölfel R. Development of a versatile and stable internal control system for RT-qPCR assays. Journal of Virological Methods. 2014; 208: 33–40. https://doi.org/10.1016/j.jviromet.2014.07.028
24. Blaise-Boisseau S., Hennechart-Collette C., Guillier L., Perelle S. Duplex real-time qRT-PCR for the detection of hepatitis A virus in water and raspberries using the MS2 bacteriophage as a process control. Journal of Virological Methods. 2010; 166 (1–2): 48–53. https://doi.org/10.1016/j.jviromet.2010.02.017
25. Borghetti I. A., Zambenedetti M. R., Requião L., Vieira D. S., Krieger M. A., de Cássia Pontello Rampazzo R. External control viral-like particle construction for detection of emergent arboviruses by real-time reverse-transcription PCR. BioMed Research International. 2019; 2019:2560401. https://doi.org/10.1155/2019/2560401
26. Wie Y., Yang C., Wie B., Huang J., Wang L., Meng S., et al. RNase-resistant virus-like particles containing long chimeric RNA sequences produced by two-plasmid coexpression system. Journal of Clinical Microbiology. 2008; 46 (5): 1734–1740. https://doi.org/10.1128/JCM.02248-07
27. Wang X., Liu F., Jiang L., Bao Y., Xiao Y., Wang H. Use of chimeric influenza viruses as a novel internal control for diagnostic rRT-PCR assays. Applied Microbiology and Biotechnology. 2016; 100 (4): 1667–1676. https://doi.org/10.1007/s00253-015-7042-y
28. Ursi J.-P., Ursi D., Ieven M., Pattyn S. R. Utility of an internal control for the polymerase chain reaction: application to detection of Mycoplasma pneumoniae in clinical specimens. Journal of Pathology, Microbiology and Immunology. 1992; 100 (7–12): 635–639. https://doi.org/10.1111/j.1699-0463.1992.tb03978.x
29. Zimmermann K., Mannhalter J. W. Technical aspects of quantitative competitive PCR. BioTechniques. 1996; 21 (2): 268–279. https://doi.org/10.2144/96212rv01
30. Brightwell G., Pearce M., Leslie D. Development of internal controls for PCR detection of Bacillus anthracis. Molecular and Cellular Probes. 1998; 12 (6): 367–377. https://doi.org/10.1006/mcpr.1998.0195
31. Zimmermann K., Rieger M., Groß P., Turecek P. L., Schwarz H. P. Sensitive single-stage PCR using custom-synthesized internal controls. BioTechniques. 2000; 28 (4): 694–702. https://doi.org/10.2144/00284st05
32. Roux G., Ravel C., Varlet-Marie E., Jendrowiak R., Bastien P., Sterkers Y. Inhibition of polymerase chain reaction: pathogen-specific controls are better than human gene amplification. PLoS ONE. 2019; 14 (9):e0219276. https://doi.org/10.1371/journal.pone.0219276
33. Liu J., Gratz J., Amour C., Nshama R., Walongo T., Maro A., et al. Optimization of quantitative PCR methods for enteropathogen detection. PLoS ONE. 2016; 11 (6):e0158199. https://doi.org/10.1371/journal.pone.0158199
34. Metlin A. E., Rybakov S. S., Gruzdev K. N., Neuvonen E., Cox J., Huovilainen A. Antigenic and molecular characterization of field and vaccine rabies virus strains in the Russian Federation. Developments in Biologicals. 2006; 125: 33–37. https://pubmed.ncbi.nlm.nih.gov/16878458
35. Jarman S. N. Amplicon: software for designing PCR primers on aligned DNA sequences. Bioinformatics. 2004; 20 (10): 1644–1645. https://doi.org/10.1093/bioinformatics/bth121
36. Sachadyn P., Kur J. The construction and use of a PCR internal control. Molecular and Cellular Probes. 1998; 12 (5): 259–262. https://doi.org/10.1006/mcpr.1998.0170
Об авторе
С. А. ЧупинРоссия
Чупин Сергей Александрович - канд. биол. наук, ведущий научный сотрудник референтной лаборатории по бешенству и BSE, ФГБУ «ВНИИЗЖ».
л. Гвардейская, 6, мкр. Юрьевец, Владимир, 600901
Рецензия
Для цитирования:
Чупин С.А. Разработка системы внутреннего контроля на основе штамма «Ла-Сота» вируса ньюкаслской болезни при диагностике бешенства методом ОТ-ПЦР. Ветеринария сегодня. 2025;14(3):249-254. https://doi.org/10.29326/2304-196X-2025-14-3-249-254
For citation:
Chupin S.A. Construction of Newcastle disease virus LaSota strain-based internal sample for rabies diagnosis with RT-PCR. Veterinary Science Today. 2025;14(3):249-254. https://doi.org/10.29326/2304-196X-2025-14-3-249-254



































